2024-07-12
한어Русский языкEnglishFrançaisIndonesianSanskrit日本語DeutschPortuguêsΕλληνικάespañolItalianoSuomalainenLatina
Involucrum GOPlot ad usum notitiarum biologicarum visualizationis adhibetur. Involucrum potius haec integrat ac visuales exprimit notitias cum eventis analysium functionum.Sed videSarcina haec ad has analyses perficiendas adhiberi non potest, solum ad eventus visualise. . In omnibus scientiis campis, difficile est res ex locorum angustiis et simplicitate ad effectum requisita realitatis describere, ideo informationes oportet ut subjiciantur et picturae ad informationes indicandas adhibeantur. Graphics bene dispositae magis notitias in spatio minori praebent. Idea sarcinae est ut utentes cito perscrutari possint magnas notitiarum copiae, trends revelare in notitiis et exemplaria et correlationes in notitia invenire.
Data visualisationi nos adiuvare possunt quaestionibus biologicis responsa invenire, hypothesin quamdam iudicare et etiam diversos angulos invenire ad diversas difficultates investigandas. Munera insidiantes huius sarcinae in structuram hierarchicam notitiarum evolvuntur, incipiendo ab altiore notitia et cum subset genesis delectorum et meatus correspondentes terminantes.
Rem cum exemplo explicemus.
Notitiam vocamus quae apud GOplot venit, quae ex GEO fluitGSE47067continens informationes transcriptome de cellis endothelialibus e duobus fibris (cerebri et cordis).Data sunt normalized et differentialiter gena expressa inveniuntur.ac deinde munus DAVID annotationis instrumentum utere (DAVID annotationem datam lente renovari et nunc non commendatur. Commendatur uti.Ire Orientem, optimum online GO locupletatio analyseos instrumentumetHaec pagina, quae analysin locupletationem efficere potest uno gradu, plus quam trecentis temporibus a CNS et aliis ante divulgatam citata est.Facere locupletationem analysi;Magister GSEA in uno articulo, super expresse tutorial) Gene annotationem genes differentialiter expressitadjusted p-value < 0.05 ) analysis ac functionis locupletationis. Haec notitia copia haec quinque genera notitiarum continet:
| nomen | describere | Data set magnitudine |
|---|---|---|
| EC$eset | Normalised gene expressio in cellulis cerebri et cordis endothelialis (3 replicat) | 20644 x 7 |
| EC$ genelist | Genera differentialiter expressa (p-value adaptata <0.05) | 2039 x 7 |
| EC$david | Proventus locupletationis functionis analysis genesis differentialis utens DAVID | 174 x 5 |
| EC$ gene | Genes et logFC | 37 x 2 |
| EC $ processum | Electus pluma vector in ditatis biologicis processibus | 7 |
Volumus videre GO vias ditatas differentialium generum expressarum, sed antequam haurire incipiamus, opus est notitias praebere quae forma requisita occurrent.In universum notitia quae ad graphum conficiendum requiruntur, a te ipso providentur, sedEst munus in hac sarcinacircle_datPotest adiuvare nos agere cum data forma。circle_datConiungere potest ad auctarium functionis analysin eventus generum delectorum et eorum logFC valorum, maxime ad genes differentialiter expressos.circle_dat Usus est valde simplex, sicut in duobus notitiis legitur. Prima notitia continet analysin functionem locupletandi, cum quattuor saltem columnis (analysis functionis ditiorationis categoria, via, gene, p-value accommodata).Secunda notitia est generum delectorum eiusque logFC, haec notitia fons esse potestlimmaEventus analysis statisticae (Nota e biographiis: Cave attendere ad duas tabulasQuomodo genes nominanturConstare, ut omnesGene symbol ). Exempla utamur ut ad formas supra scriptas spectant.
- #安装已发布的稳定版本
- #install.packages('GOplot')
- #安装github上的开发版本
- #install_github('wencke/wencke.github.io')
- #载入包
- library(GOplot)
- #读入包内自带的数据
- data(EC)
- #查看功能富集分析结果的数据格式
- head(EC$david)
-
- ## Category ID Term
- ## 1 BP GO:0007507 heart development
- ## 2 BP GO:0001944 vasculature development
- ## 3 BP GO:0001568 blood vessel development
- ## 4 BP GO:0048729 tissue morphogenesis
- ## 5 BP GO:0048514 blood vessel morphogenesis
- ## 6 BP GO:0051336 regulation of hydrolase activity
- ## Genes
- ## 1 DLC1, NRP2, NRP1, EDN1, PDLIM3, GJA1, TTN, GJA5, ZIC3, TGFB2, CERKL, GATA6, COL4A3BP, GAB1, SEMA3C, MKL2, SLC22A5, MB, PTPRJ, RXRA, VANGL2, MYH6, TNNT2, HHEX, MURC, MIB1, FOXC2, FOXC1, ADAM19, MYL2, TCAP, EGLN1, SOX9, ITGB1, CHD7, HEXIM1, PKD2, NFATC4, PCSK5, ACTC1, TGFBR2, NF1, HSPG2, SMAD3, TBX1, TNNI3, CSRP3, FOXP1, KCNJ8, PLN, TSC2, ATP6V0A1, TGFBR3, HDAC9
- ## 2 GNA13, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, FOXO1, GJA5, TGFB2, WARS, CERKL, APOE, CXCR4, ANG, SEMA3C, NOS2, MKL2, FGF2, RAPGEF1, PTPRJ, RECK, EFNB2, VASH1, PNPLA6, THY1, MIB1, NUS1, FOXC2, FOXC1, CAV1, CDH2, MEIS1, WT1, CDH5, PTK2, FBXW8, CHD7, PLCD1, PLXND1, FIGF, PPAP2B, MAP2K1, TBX4, TGFBR2, NF1, TBX1, TNNI3, LAMA4, MEOX2, ECSCR, HBEGF, AMOT, TGFBR3, HDAC7
- ## 3 GNA13, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, FOXO1, GJA5, TGFB2, WARS, CERKL, APOE, CXCR4, ANG, SEMA3C, NOS2, MKL2, FGF2, RAPGEF1, PTPRJ, RECK, VASH1, PNPLA6, THY1, MIB1, NUS1, FOXC2, FOXC1, CAV1, CDH2, MEIS1, WT1, CDH5, PTK2, FBXW8, CHD7, PLCD1, PLXND1, FIGF, PPAP2B, MAP2K1, TBX4, TGFBR2, NF1, TBX1, TNNI3, LAMA4, MEOX2, ECSCR, HBEGF, AMOT, TGFBR3, HDAC7
- ## 4 DLC1, ENAH, NRP1, PGF, ZIC2, TGFB2, CD44, ILK, SEMA3C, RET, AR, RXRA, VANGL2, LEF1, TNNT2, HHEX, MIB1, NCOA3, FOXC2, FOXC1, TGFB1I1, WNT5A, COBL, BBS4, FGFR3, TNC, BMPR2, CTNND1, EGLN1, NR3C1, SOX9, TCF7L1, IGF1R, FOXQ1, MACF1, HOXA5, BCL2, PLXND1, CAR2, ACTC1, TBX4, SMAD3, FZD3, SHANK3, FZD6, HOXB4, FREM2, TSC2, ZIC5, TGFBR3, APAF1
- ## 5 GNA13, CAV1, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, CDH2, MEIS1, WT1, TGFB2, WARS, PTK2, CERKL, APOE, CXCR4, ANG, SEMA3C, PLCD1, NOS2, MKL2, PLXND1, FIGF, FGF2, PTPRJ, TGFBR2, TBX4, NF1, TBX1, TNNI3, PNPLA6, VASH1, THY1, NUS1, MEOX2, ECSCR, AMOT, HBEGF, FOXC2, FOXC1, HDAC7
- ## 6 CAV1, XIAP, AGFG1, ADORA2A, TNNC1, TBC1D9, LEPR, ABHD5, EDN1, ASAP2, ASAP3, SMAP1, TBC1D12, ANG, TBC1D14, MTCH1, TBC1D13, TBC1D4, TBC1D30, DHCR24, HIP1, VAV3, NOS1, NF1, MYH6, RICTOR, TBC1D22A, THY1, PLCE1, RNF7, NDEL1, CHML, IFT57, ACAP2, TSC2, ERN1, APAF1, ARAP3, ARAP2, ARAP1, HTR2A, F2R
- ## adj_pval
- ## 1 0.000002170
- ## 2 0.000010400
- ## 3 0.000007620
- ## 4 0.000119000
- ## 5 0.000720000
- ## 6 0.001171166
-
- #查看基因的数据格式
- head(EC$genelist)
-
- ## ID logFC AveExpr t P.Value adj.P.Val B
- ## 1 Slco1a4 6.645388 1.2168670 88.65515 1.32e-18 2.73e-14 29.02715
- ## 2 Slc19a3 6.281525 1.1600468 69.95094 2.41e-17 2.49e-13 27.62917
- ## 3 Ddc 4.483338 0.8365231 65.57836 5.31e-17 3.65e-13 27.18476
- ## 4 Slco1c1 6.469384 1.3558865 59.87613 1.62e-16 8.34e-13 26.51242
- ## 5 Sema3c 5.515630 2.3252117 58.53141 2.14e-16 8.81e-13 26.33626
- ## 6 Slc38a3 4.761755 0.9218670 54.11559 5.58e-16 1.76e-12 25.70308
Post intellectum initus notitiarum duarum formarum, uti potescirlce_datmunus generandi extractionem data.
- # 生成画图所需的数据格式
- circ <- circle_dat(EC$david, EC$genelist)
-
- head(circ)
-
- ## category ID term count genes logFC adj_pval
- ## 1 BP GO:0007507 heart development 54 DLC1 -0.9707875 2.17e-06
- ## 2 BP GO:0007507 heart development 54 NRP2 -1.5153173 2.17e-06
- ## 3 BP GO:0007507 heart development 54 NRP1 -1.1412315 2.17e-06
- ## 4 BP GO:0007507 heart development 54 EDN1 1.3813006 2.17e-06
- ## 5 BP GO:0007507 heart development 54 PDLIM3 -0.8876939 2.17e-06
- ## 6 BP GO:0007507 heart development 54 GJA1 -0.8179480 2.17e-06
- ## zscore
- ## 1 -0.8164966
- ## 2 -0.8164966
- ## 3 -0.8164966
- ## 4 -0.8164966
- ## 5 -0.8164966
- ## 6 -0.8164966
circObiectum habet notitias octo columnas, scilicet
categoria: BP (Processus Biologicus), CC (Component Cellular) vel MF (Molecular Function)
ID: GO id (columna ad libitum si vis instrumento analysi functionis uti quod in GO id non fundatur, columnam ID eligere non potes; ID hic etiam KEGG ID esse potest)
terminus: GO viam
numerare: numerus genes in unaquaque via
gene: gene name - logFC: logFC cuiusque gene
adj_pval: p valorem accommodatum, meatus cum adj_pval<0.05 insigniter locupletatus
zscore: zscore non refert ad methodum statisticam ordinationem, sed faciliorem calculi valorem aestimandi num processus biologicus (/munus hypotheticus/cellularis) magis decrescere (valorem negativum) vel incrementum (valorem positivum).Methodus calculi est numerus genesis erecti minus ordinatus numerus genesis sub quadrata radice numeri genesis in quaque via divisus.
Cum primum notitias inspicimus, vias quam plurimas e graphio demonstrare cupimus, et etiam pretiosas vias invenire volumus, ut aliquot parametris ad momentum aestimandum velimus. Bar chartulae saepe ad exemplum datae describendae sunt, ergo munus GOBar uti possumus ut chartulam speciosam cito efficiam.
Primum, simplex talea chart directe generaturGO TermssecundumzscoreSort vectes;-log(adj p-value); Color significatzscore, blue indicatz-scorevalorem negativum, gene expressio in via correspondente verisimilius est decrescere, indicata in rubeoz-score valorem positivum est, gene expressio in via correspondente verisimilius auget. Si vis, ordo mutari potest per ordinem parametri constituendo. zscore ad FALSUS, quo in casu vectes ordinantur secundum eorum significationem.
- # 生成简单的条形图
- GOBar(subset(circ, category == 'BP'))
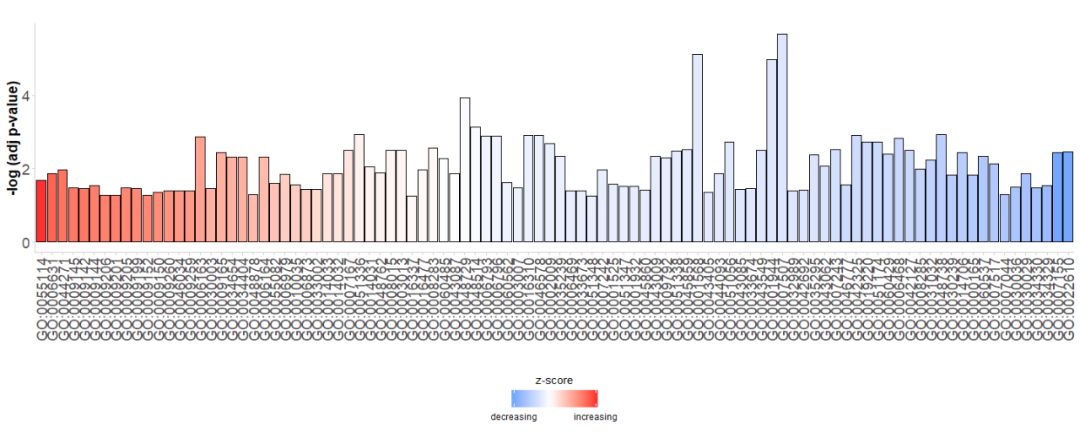
#GOBar(subset(circ, category == 'BP',order.by.zscore=FALSE))Praeterea modulum ostentationis muta ut chart vectem trahat secundum categoriam canalis.
- #根据通路的类别来绘制条形图
- GOBar(circ, display = 'multiple')
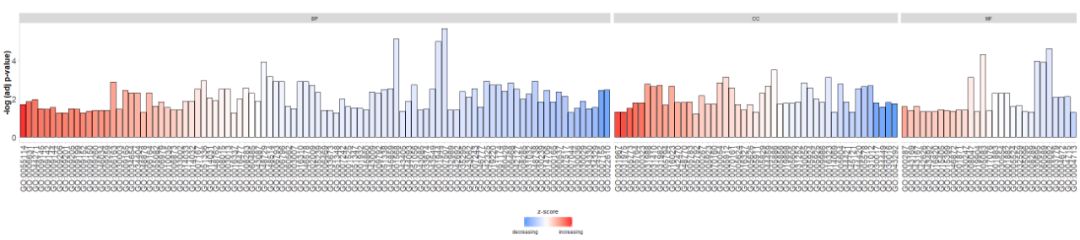
Adde titulum et usum parametrizsc.colMutatiozscorecolore s.
- # Facet the barplot, add a title and change the colour scale for the z-score
- GOBar(circ, display = 'multiple', title = 'Z-score coloured barplot', zsc.col = c('yellow', 'black', 'cyan'))
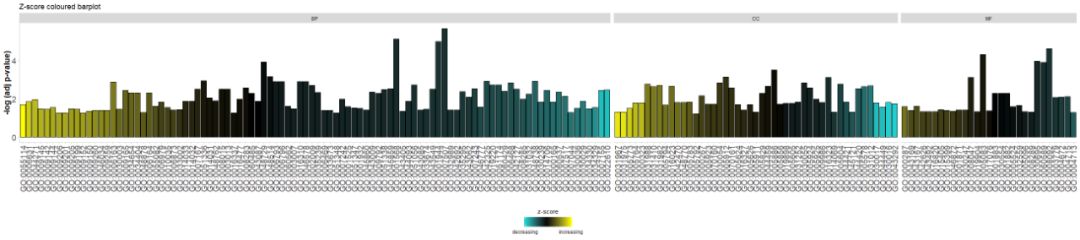
Bar chartulae usitatissimae sunt et facile ad intellegendum, sed chartis bullae uti possumus ad informationem de notitia magis proponendam.
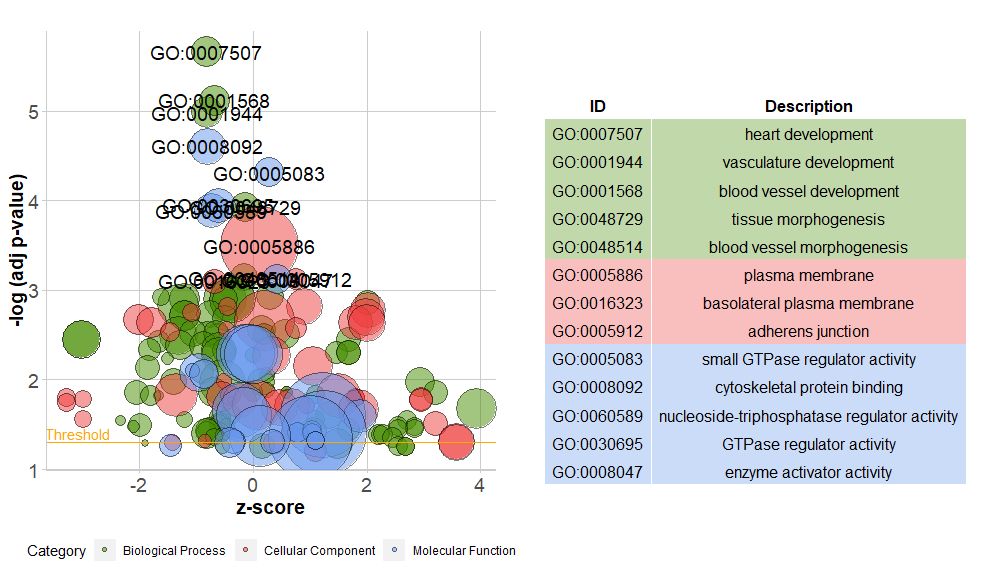
Axis horizontalis estzscore; Axis verticalis est-log(adj p-value), ut superior chart talea , quo altior est , insignior ditior est ;circ$count color correspondens categoriae viae correspondens, viridis est processus biologicus, ruber est pars cellulosa, et munus hypotheticum caeruleum est.Potest intravit per?GOBubble GOBubble vide paginam adiuvantis functionis mutare omnes ambitum imaginis. Defalta, uterque circulus cum GO ID respondente signatur, et tabula ostendens debitam relationem inter terminum GO ID et GO etiam in dextro monstratur.Morbi laoreet posuere pertable.legendfor*FALSE occultare. Si viam descriptionis ostendere vis, parametrum ID FALSUS pone.Attamen ob spatium limitatum et circulis imbricatis, non omnes circuli signantur, solum-log(adj p-value) > 3(default is 5).
- # 生成泡泡图,并展示-log(adj p-value) > 3 的通路的GO ID
- GOBubble(circ, labels = 3)
Si titulum addere vis chartae bullae, vel colorem circuli denotare et meatus cuiusque praedicamenti separatim monstrare et in limine monstratum GO ID limine mutare, sequentes parametros addere potes:
GOBubble(circ, title = 'Bubble plot', colour = c('orange', 'darkred', 'gold'), display = 'multiple', labels = 3)
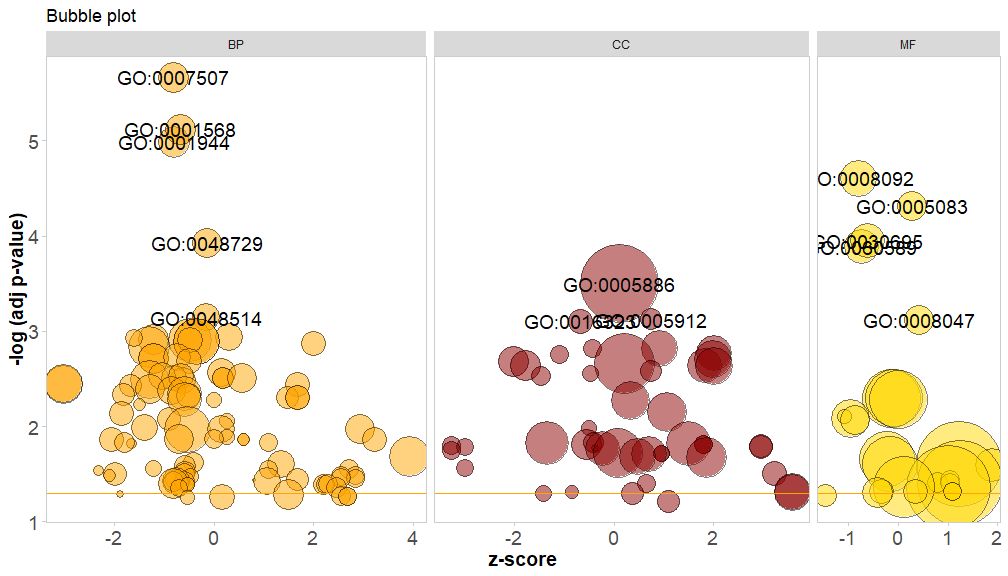
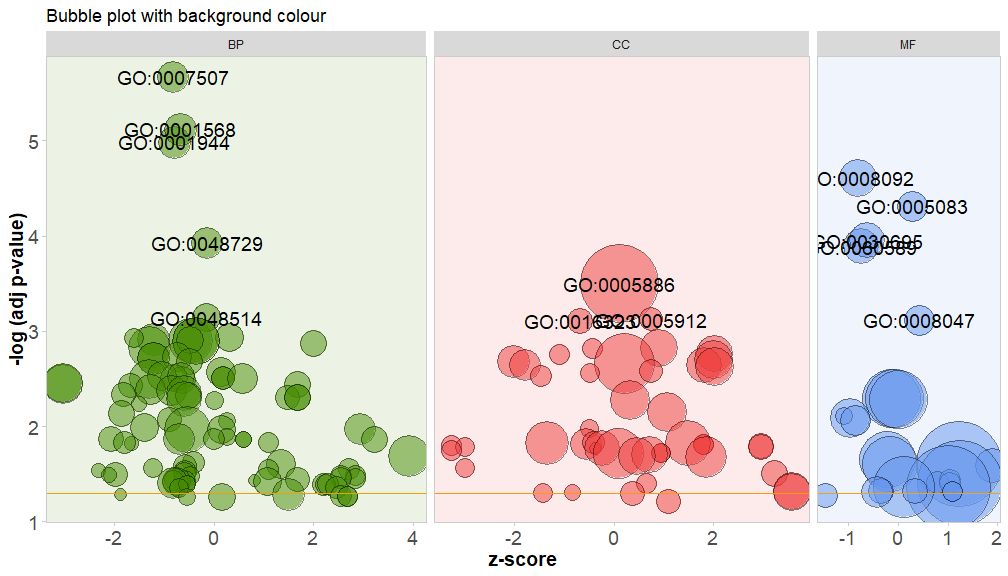
Color subiectum ordinis canalis ponendo modulum bg.col ad VERA.
GOBubble(circ, title = 'Bubble plot with background colour', display = 'multiple', bg.col = T, labels = 3)
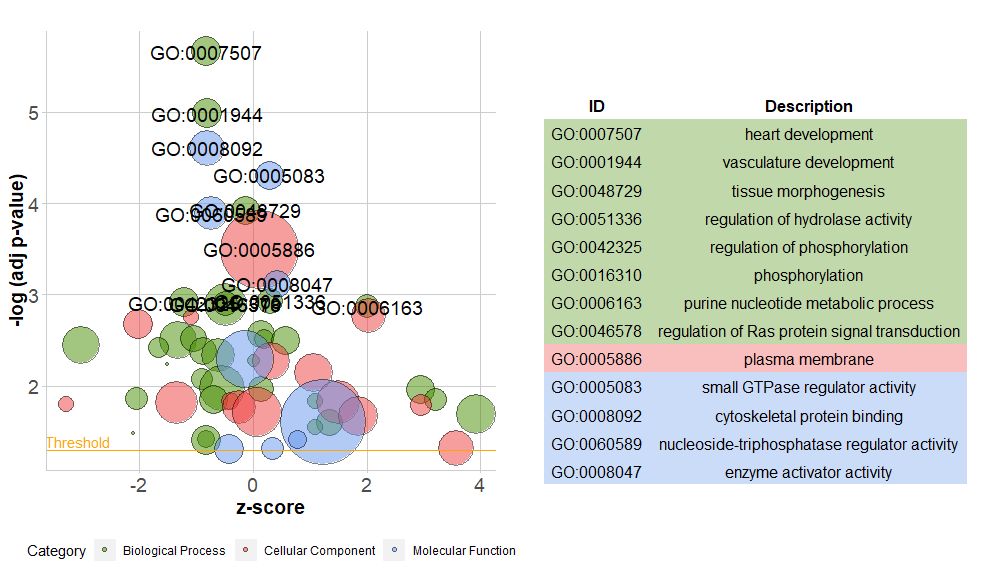
Nova versio in sarcina novum munus continetreduce_overlap Hoc munus potest reducere numerum rerum superuacentium, id est, omnes vias delere, quarum gena aliudque maius est quam vel aequale limine statuto, et unum tramitem ab unoquoque coetum retinet tantum repraesentativum, cuiuscumque ostentationis omnium. meatus in GO. Reducendo numerum vocabulorum superuacentium, facilitas insidiarum (ut bullae machinae) insigniter emendatur.
- # reduce_overlap,参数设置为0.75
- reduced_circ <- reduce_overlap(circ, overlap = 0.75)
-
- GOBubble(reduced_circ, labels = 2.8)
Etsi grapha ostendens omnia informationes adiuvari potest ut detegant quae maxime significantia itinera sunt, res tamen pendet ex hypothesibus et notionibus, quas cum notitia confirmare vis, et maximae vias non necessario possunt esse quos interest. Ideo in manually eligendo perutile meatum (EC$process ) schemate opus est ut accuratiorem nobis informationem de hoc certo limitum statuto ostendat.Sed oritur quaestio has figuras exhibere: interdum difficile est interpretarizscore Informationis dedi.Ceterum methodus haec calculi non est universalis. Ut supra ostensum est, simpliciter numerus genesis regularis minus ordinatus numerus genesis per radicem quadratam numeri genesis in quaque via divisus est, utens.GOCircleConsequens graph etiam hoc inculcat.
Circulus exterior circuli diagramma demonstrat logFC genes cuiuslibet semitae sicut puncta dispersa. Red circuli upregulation et caerulei indicant downregulation.Morbi potest esselfc.col Color mutabilis. Inde etiam est quod in aliquibus casibus maximi momenti meatus zscores habent prope nullas. Nulla a zscore non id, varius purus. Hoc modo ostendit zscore mensurae asperae esse, quia patet etiam zscore non rationem habere dependentiae functionis et activationis singulorum generum in processibus biologicis.
GOCircle(circ)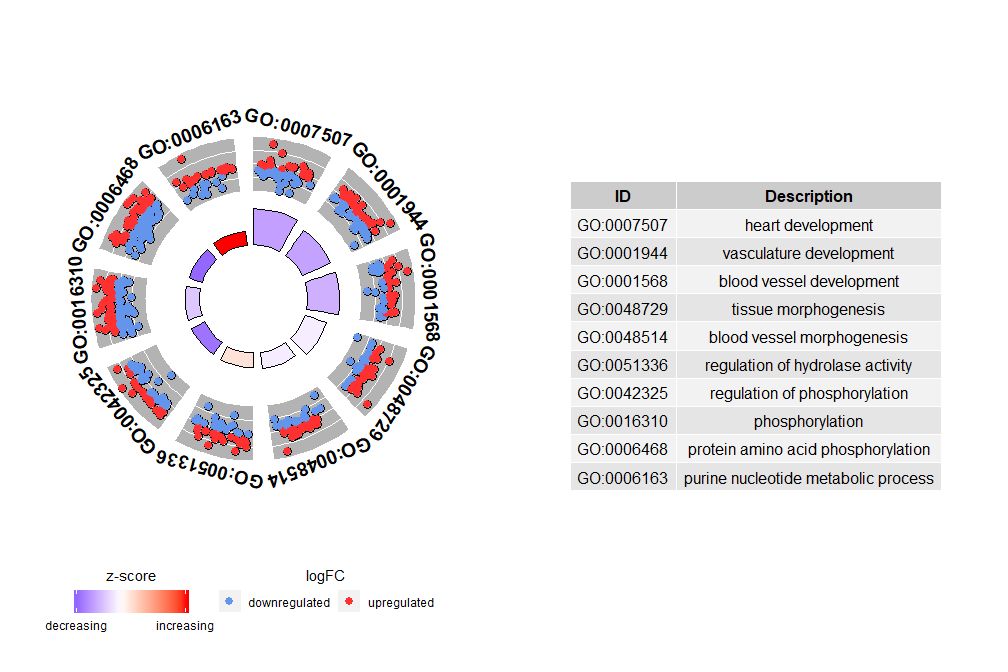
nsub Parametri numeri seu character vectores erigi possunt. Si vector character est, GO ID vel iter ad ostentationem continet;
- # 生成特定通路的圈图
- IDs <- c('GO:0007507', 'GO:0001568', 'GO:0001944', 'GO:0048729', 'GO:0048514', 'GO:0005886', 'GO:0008092', 'GO:0008047')
- GOCircle(circ, nsub = IDs)
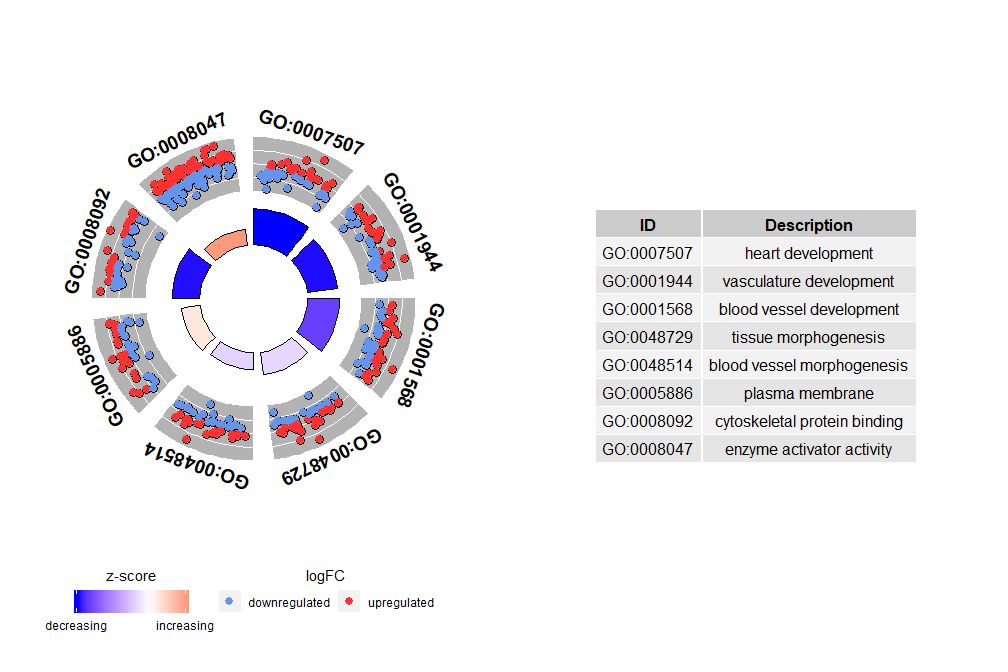
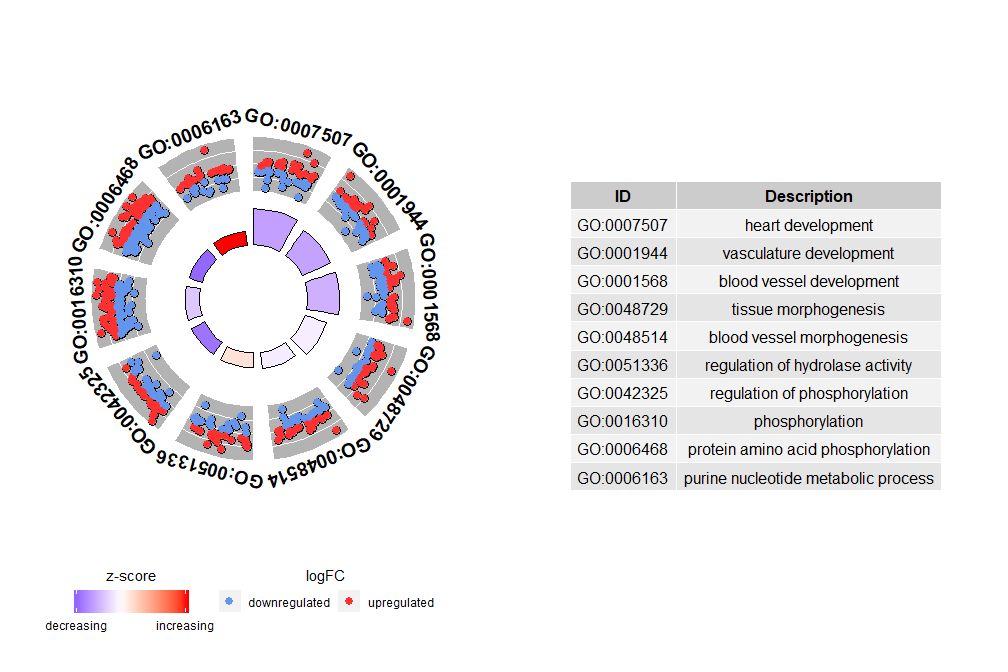
Si nsub vector numericus est, numerus definit numerum ut ostendat. Incipit a primo versu initus notitiae artis. Haec visualisatio solum laborat cum minori notitia. Maximus numerus defaltis canalibus ad XII. Etsi numerus canalium reducitur, copia informationum augetur.
- # 圈图展示数据前十个通路
- GOCircle(circ, nsub = 10)
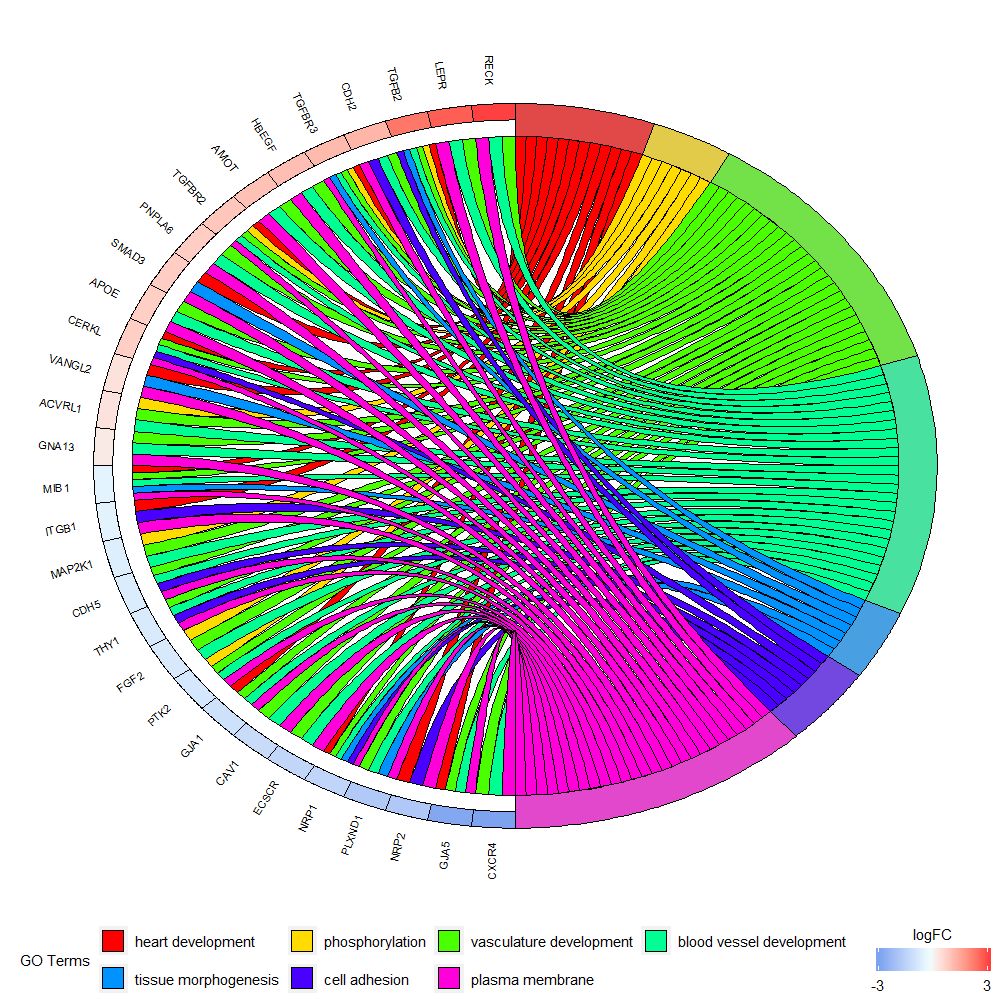
GOChord relationem inter selectas genes et vias et genesis logFC ostendere potest.Primum matrix inicere debes, quam te ipsum aedificare potes0-1Matrix, functionibus uti poteschord_dat Construe. Hoc munus tres parametri habet: notitia, genes et processus, e quibus duo postremi parametri saltem unum modulum habere debent.Tum munuscircle_datExpressionem datam coniunge cum proventus ex analysibus functionis.
Bar chartis et chartis bullae primum impressionem notitiarum tibi dare possunt. Nunc, gena quaedam et vias quas aestimamus pretiosas eligere potes de relationibus inter genes et plures vias. Num quaedam genera multiplicibus processibus iuncta sint, haud facile est inspicere. GOChord supplet pro defectibus GOCircle. Ordines generati datae sunt genes et columnae sunt semitae.
- # 找到感兴趣的的基因,这里我们以EC$genes为例
- head(EC$genes)
-
- ## ID logFC
- ## 1 PTK2 -0.6527904
- ## 2 GNA13 0.3711599
- ## 3 LEPR 2.6539788
- ## 4 APOE 0.8698346
- ## 5 CXCR4 -2.5647537
- ## 6 RECK 3.6926860
-
- # 获得感兴趣基因的通路
- EC$process
-
- ## [1] "heart development" "phosphorylation"
- ## [3] "vasculature development" "blood vessel development"
- ## [5] "tissue morphogenesis" "cell adhesion"
- ## [7] "plasma membrane"
-
- # 使用chord_dat构建矩阵
- chord <- chord_dat(circ, EC$genes, EC$process)
- head(chord)
-
- ## heart development phosphorylation vasculature development
- ## PTK2 0 1 1
- ## GNA13 0 0 1
- ## LEPR 0 0 1
- ## APOE 0 0 1
- ## CXCR4 0 0 1
- ## RECK 0 0 1
- ## blood vessel development tissue morphogenesis cell adhesion
- ## PTK2 1 0 0
- ## GNA13 1 0 0
- ## LEPR 1 0 0
- ## APOE 1 0 0
- ## CXCR4 1 0 0
- ## RECK 1 0 0
- ## plasma membrane logFC
- ## PTK2 1 -0.6527904
- ## GNA13 1 0.3711599
- ## LEPR 1 2.6539788
- ## APOE 1 0.8698346
- ## CXCR4 1 -2.5647537
- ## RECK 1 3.6926860
In exemplo, duos parametros transivimus. Si modulus genesis tantum designatus est, effectus est index genesis et omnes constructiones processus cum uno saltem gene specifico.0-1matrix;processparametri, effectus est ut omnia genesis generant0-1 Matrix generum unum saltem processum in indice assignatum est. Scias modo speciem genesis et processus parametri in amplissima matrice 0-1 provenire, inde in confusione visualizationis proventuum.
- head(circ)
-
- ## category ID term count genes logFC adj_pval
- ## 1 BP GO:0007507 heart development 54 DLC1 -0.9707875 2.17e-06
- ## 2 BP GO:0007507 heart development 54 NRP2 -1.5153173 2.17e-06
- ## 3 BP GO:0007507 heart development 54 NRP1 -1.1412315 2.17e-06
- ## 4 BP GO:0007507 heart development 54 EDN1 1.3813006 2.17e-06
- ## 5 BP GO:0007507 heart development 54 PDLIM3 -0.8876939 2.17e-06
- ## 6 BP GO:0007507 heart development 54 GJA1 -0.8179480 2.17e-06
- ## zscore
- ## 1 -0.8164966
- ## 2 -0.8164966
- ## 3 -0.8164966
- ## 4 -0.8164966
- ## 5 -0.8164966
- ## 6 -0.8164966
-
- # Generate the matrix with a list of selected genes
- chord_genes <- chord_dat(data = circ, genes = EC$genes)
- head(chord_genes)
-
- ## heart development vasculature development blood vessel development
- ## PTK2 0 1 1
- ## GNA13 0 1 1
- ## LEPR 0 1 1
- ## APOE 0 1 1
- ## CXCR4 0 1 1
Haec chartula intenditur ostendere minorem subsetarium altae dimensivarum notitiarum. Parametri sunt duo maxime qui accommodentur;gene.orderetnlfc . genes parametri specificari possunt sicut 'logFC', 'alphabeticum', 'nullus'. Re quidem vera parametrum genesis gene ut logFC generaliter definimus; parameter nlfc est unus ex praecipuis parametris huius functionis, quia tractare potest quomodo unaquaeque gene 0 vel plures valores logFC in matrice praesentes habeat. Ergo parametri notandae sunt ad vitandos errores.
Exempli gratia, si vulvam habes sine valoribus logFC, pone debesnlfc=0 Vel analysin differentialem expressionem in genes per plures conditiones vel batches faciendas. In hoc casu, unaquaeque gena continet multiplices valores logFC, et numerus columnae nlfc=logFC ponendus est. Valor default "I" est quia creditur frequentius tempus unum tantum valorem logFC per gene fore. Utere spatii parametri ad definiendum spatium inter rectangula colorata logFC repraesentantia. Gen.size parameter significat magnitudinem fontis nominis gene, et gene. Spatium significat magnitudinem spatii inter nomina gene.
- chord <- chord_dat(data = circ, genes = EC$genes, process = EC$process)
- GOChord(chord, space = 0.02, gene.order = 'logFC', gene.space = 0.25, gene.size = 5)
-
- ## Warning: Using size for a discrete variable is not advised.
-
- ## Warning: Removed 7 rows containing missing values (geom_point).
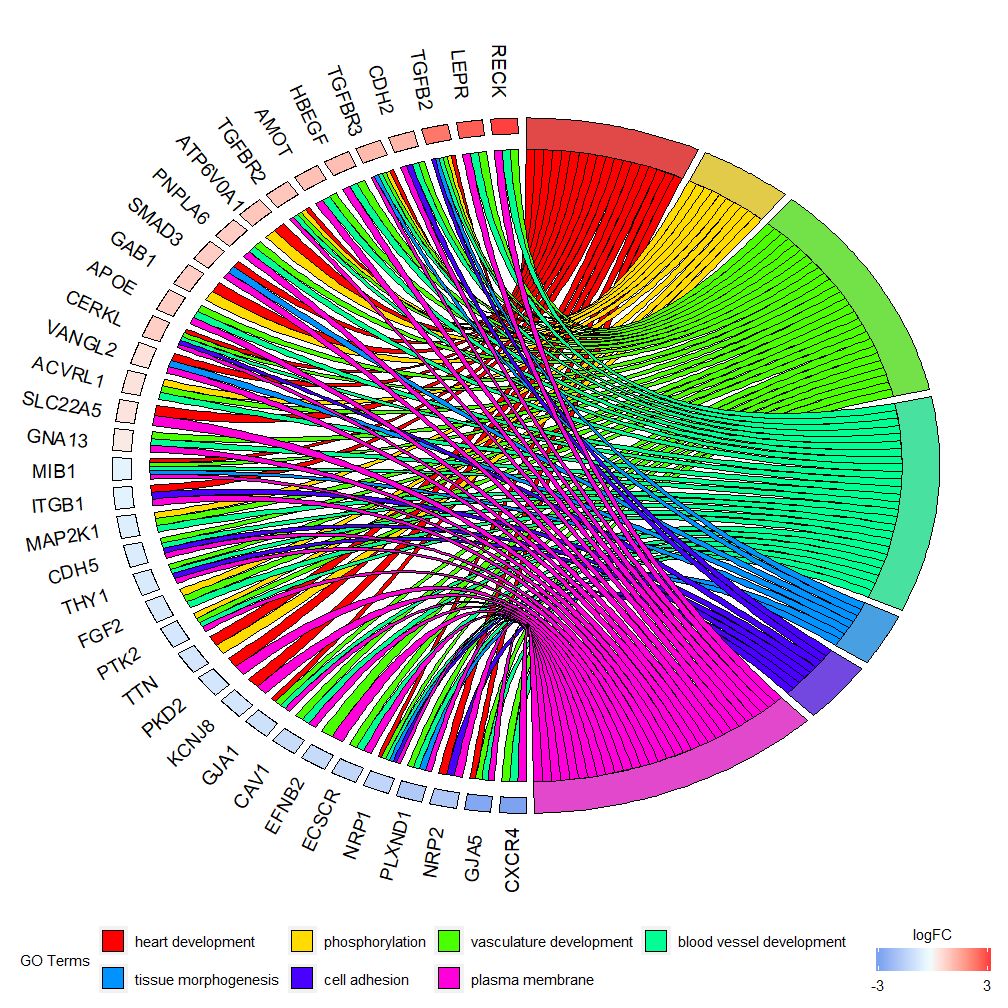
Poni potest secundum valorem logFCgene.order=‘logFC’ gena secundum valores eorum logFC disponuntur. Interdum imago aliquantulum conferta fieri potest et hoc automari potest utendo limite parametri ad numerum generum vel viae ostensorum minuendum. Terminus vector est cum duobus valoribus intervallis (defalta est c(0,0)). Valor prima designat numerum minimorum meatum quibus gena assignanda est. Secundum valorem determinatum numerum genesis ad semitam assignatum.
- # 仅显示分配给至少三个通路的基因
- GOChord(chord, limit = c(3, 0), gene.order = 'logFC')
-
- ## Warning: Using size for a discrete variable is not advised.
-
- ## Warning: Removed 7 rows containing missing values (geom_point).
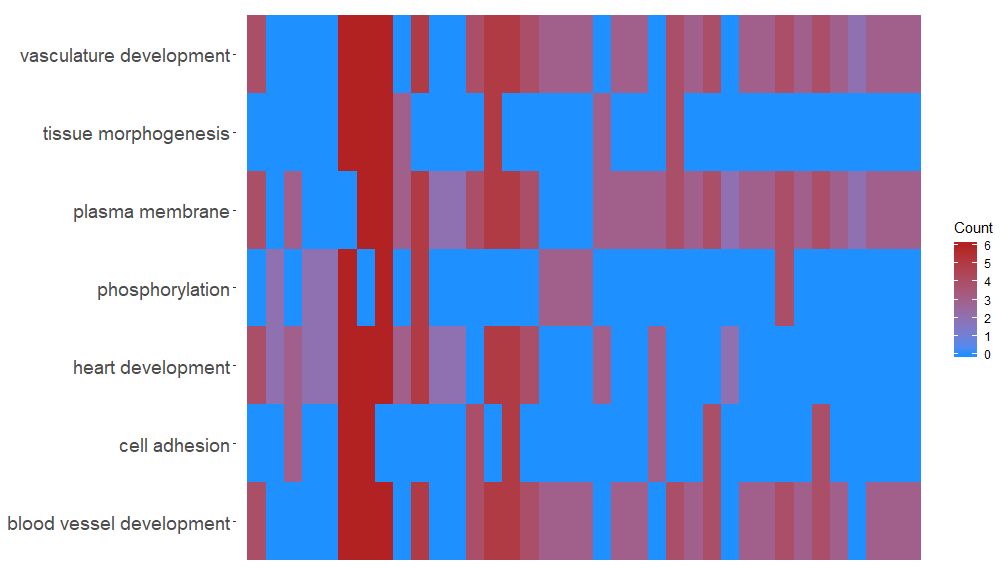
Munus GOHeat relationem inter gena et vias uti potest in tabula calefacta, similis GOChord, ostendere. Processus biologici horizontaliter monstrantur et gena verticaliter monstrantur. Unaquaeque columna dividitur in rectangula parva, et color plerumque in valore logFC dependet. Praeterea genes locupletati similium meatus muneris aggregati erant. Duplex modus est color lectionis heatmap, secundum ambitum nlfc. Si nlfc = 0, color est numerus meatus ditatur pro unaquaque gene. Exempla vide in singulis:
- # First, we use the chord object without logFC column to create the heatmap
- GOHeat(chord[,-8], nlfc = 0)
GOHeat(chord[,-8])Color respondet logFC gene in casu nlfc = 1
GOHeat(chord, nlfc = 1, fill.col = c('red', 'yellow', 'green'))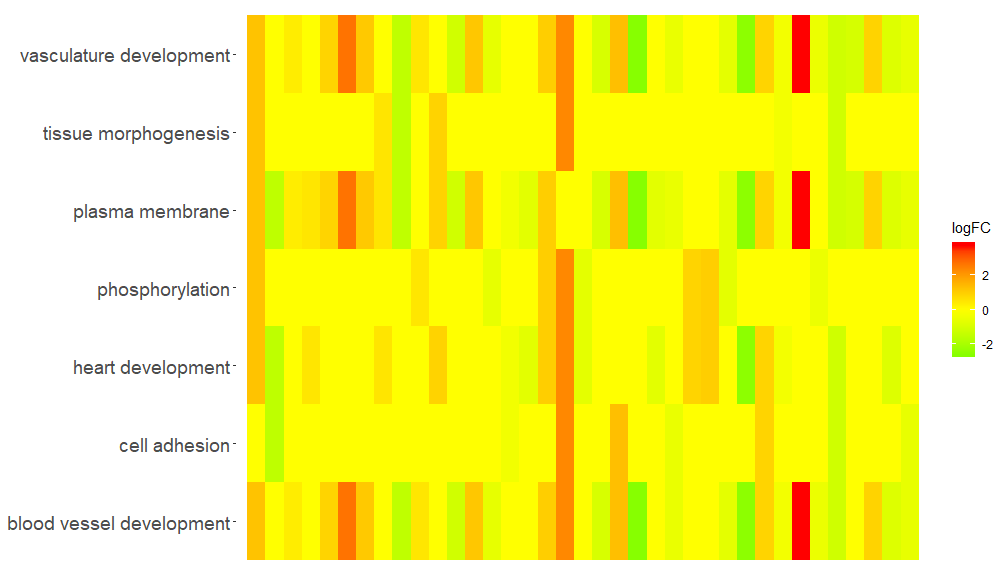
Idea post GOClustris functionis est quam maxime notitias proponere. Hic est exemplum.
- GOCluster(circ, EC$process, clust.by = 'logFC', term.width = 2)
-
- ## Warning: Using size for a discrete variable is not advised.
-
- ## Warning: Removed 7 rows containing missing values (geom_point).
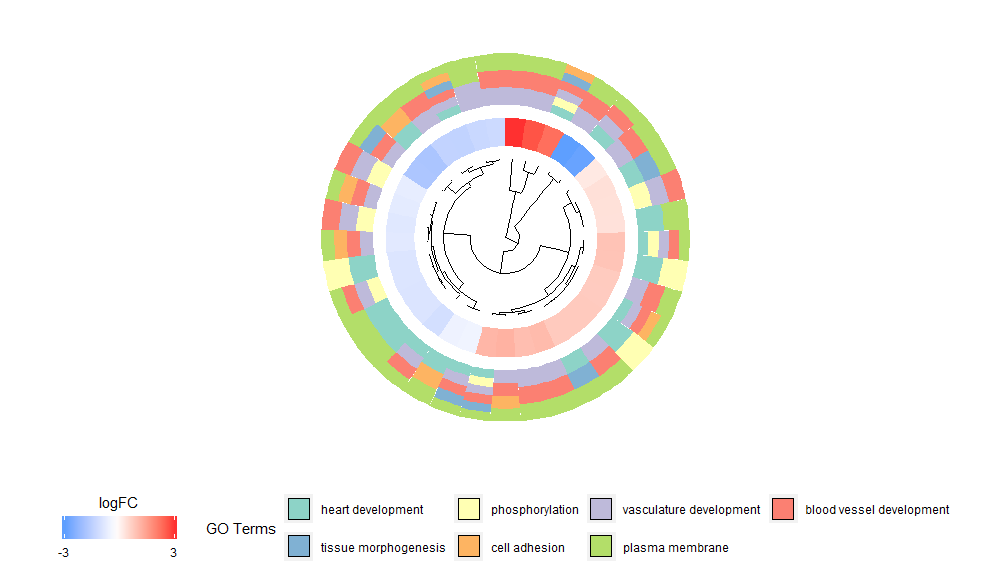
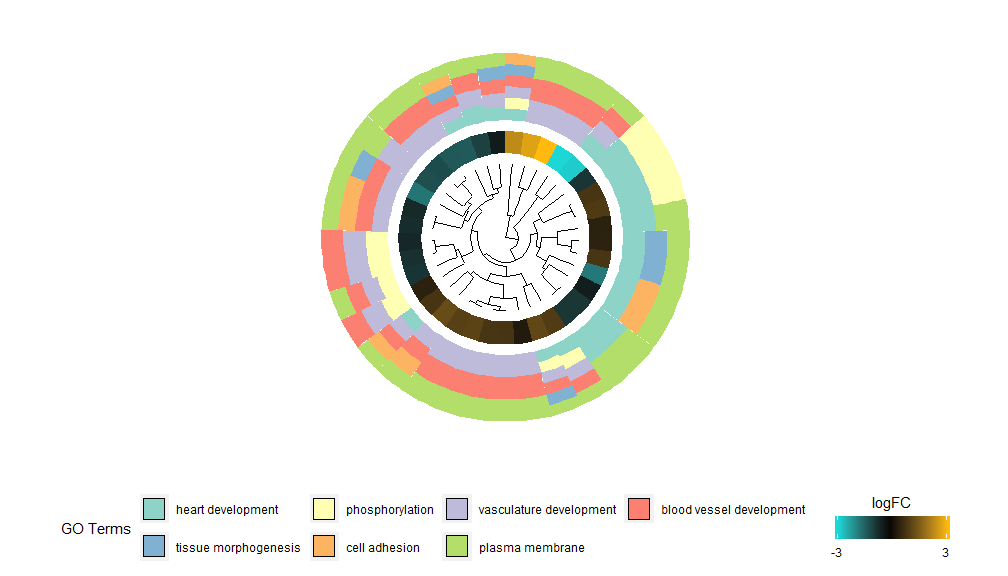
Conglobatio hierarchica est popularis analysis insuibilis conglobatio methodi pro expressione gene, quae generat generum conglobationem cum expressio exemplari, ita ut ligaturas multiplices coetus coordinatarum vel officiatis genesis contineat. GOCluster utitur adhclust Methodus hierarchicas condensationes generum expressionum permittit. Si vis distantiam metricam vel ligaturam algorithmum mutare, parametris metricis et glomis utere respective. Elige circulationem extensionem ut dictum est, non solum efficax, sed etiam visibiliter appellans. Primus circulus iuxta dendrographum logFC generum significat, quod est actu folium arboris pampinei. Si interest in multiplicibus oppositionibus modulum nlfc mutare potes, per defaltam ad "1" adponitur, ut unus tantum anulus ducatur. Valores logFC sunt color-coded utentes scale color usoris definibilis (lfc.col); Ut melius inspicias, numerus canalium redactus est, et color canalium utendo modulo termino.adhuc praesto?GOCluster videre quam mutare ambitum. Praecipuus modulus huius functionis est cluster.by, qui gregari potest per exemplaria expressionis gene ('logFC', ut supra demonstratum est) vel genera functionis ('verbis') distingui potest.
- GOCluster(circ, EC$process, clust.by = 'term', lfc.col = c('darkgoldenrod1', 'black', 'cyan1'))
-
- ## Warning: Using size for a discrete variable is not advised.
-
- ## Warning: Removed 7 rows containing missing values (geom_point).
Venn schemata adhiberi possunt ad relationes deprehendendas inter varios indices genesis differentialiter expressos, vel ad explorandum intersectionem multiplex viae viae in analysibus functionis. Venn schemata non solum numerum generum imbricatis monstrant, sed etiam informationem de exemplaris expressionis gene (plerumque sursum ordinato, saepe deorsum ordinato vel contra regulato). In statu, usque ad tres datasets input ponuntur. Notitia initus continet duas saltem columnas: unam pro nominibus gene et unam pro valores logFC.
- l1 <- subset(circ, term == 'heart development', c(genes,logFC))
- l2 <- subset(circ, term == 'plasma membrane', c(genes,logFC))
- l3 <- subset(circ, term == 'tissue morphogenesis', c(genes,logFC))
- GOVenn(l1,l2,l3, label = c('heart development', 'plasma membrane', 'tissue morphogenesis'))
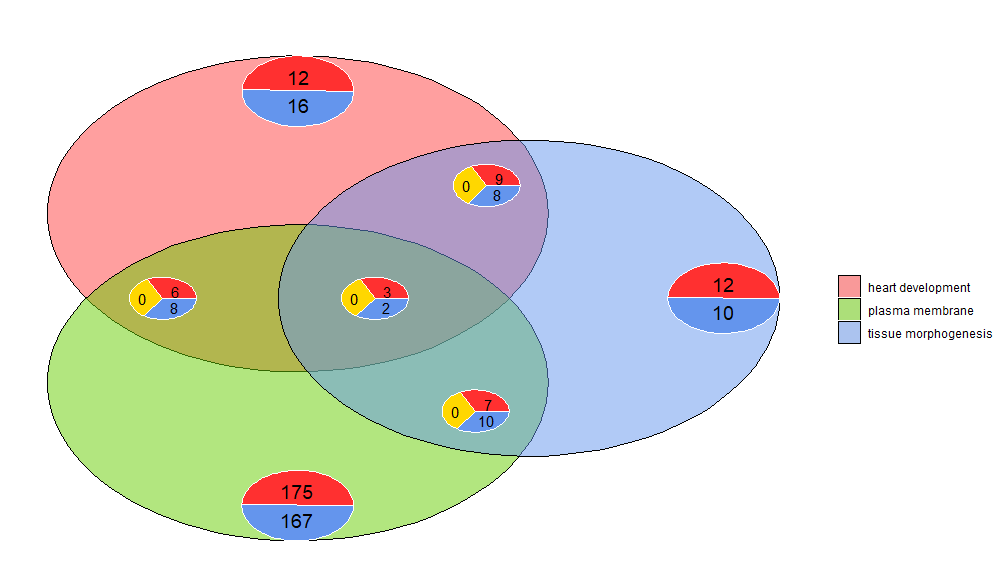
Exempli causa, progressio cardiaca et textus morphogenesis 22 genes habent, 12 sursum ordinantur et 10 deorsum ordinantur. Magni interest notandum est chartulas scriblitas non ostendunt notitias redundantes. Si ergo tres notitiae copiae comparentur, genes communis omnibus notitiae (in chartis medii pie) in aliis chartis scriblitae non comprehenduntur. Hoc instrumentum in shinyapp https://wwalter.shinyapps.io/Venn/ praesto est, instrumentum interactivum magis interactivum est, circulus aream proportionalem habet numero genae in dataset, et lapsus adhiberi potest ad movendum. chartula chartula parva, et GOVenn Features habet omnia bene mutandi extensionem argumenti ac etiam imagines et tabulas gene.
Software paginam prore: https://wencke.github.io/