2024-07-12
한어Русский языкEnglishFrançaisIndonesianSanskrit日本語DeutschPortuguêsΕλληνικάespañolItalianoSuomalainenLatina
जैविकदत्तांशस्य दृश्यीकरणाय GOPlot संकुलस्य उपयोगः भवति । अपितु एतत् संकुलं कार्यात्मकविश्लेषणस्य परिणामैः सह अभिव्यक्तिदत्तांशं एकीकृत्य दृश्यमानं करोति ।परन्तु सावधानाः भवन्तुएतानि विश्लेषणं कर्तुं एतत् संकुलं उपयोक्तुं न शक्यते, केवलं परिणामानां दृश्यीकरणार्थम् । . विज्ञानस्य सर्वेषु क्षेत्रेषु स्थानस्य बाधायाः कारणात्, परिणामानां कृते आवश्यकस्य सरलतायाः च कारणेन वस्तुनां यथार्थरूपेण वर्णनं कठिनं भवति, अतः सूचनानां दृश्यीकरणं, सूचनानां प्रसारणार्थं चित्राणां उपयोगः च आवश्यकः सुविकसितानि चित्राणि न्यूने स्थाने अधिकानि सूचनानि ददति । संकुलस्य विचारः अस्ति यत् उपयोक्तारः शीघ्रं बृहत् परिमाणेन दत्तांशस्य परीक्षणं कर्तुं, दत्तांशेषु प्रवृत्तिः प्रकाशयितुं, दत्तांशेषु प्रतिमानं सहसम्बन्धं च अन्वेष्टुं समर्थाः भवेयुः
दत्तांशदृश्यीकरणं जैविकप्रश्नानां उत्तराणि अन्वेष्टुं, कस्यापि परिकल्पनायाः न्यायं कर्तुं, भिन्नसमस्यानां अन्वेषणार्थं भिन्नकोणानां आविष्कारं कर्तुं अपि साहाय्यं कर्तुं शक्नोति । तथा च अस्य संकुलस्य प्लॉटिङ्गकार्यं दत्तांशस्य श्रेणीबद्धसंरचनायाः आधारेण विकसितं भवति, समग्रदत्तांशतः आरभ्य चयनितजीनानां उपसमूहेन तदनुरूपमार्गेण च समाप्तं भवति
उदाहरणेन सह ठोसरूपेण व्याख्यास्यामः।
वयं GOplot इत्यनेन सह यत् दत्तांशं आगच्छति तत् वदामः, यत् GEO इत्यस्मात् आगच्छतिGSE47067, यस्मिन् द्वयोः ऊतकयोः (मस्तिष्कं हृदयं च) अन्तःस्थकोशिकानां ट्रांसक्रिप्टोम-सूचना भवतिदत्तांशः सामान्यीकृतः भवति तथा च भिन्नरूपेण अभिव्यक्ताः जीनाः प्राप्यन्ते ।, ततः DAVID फंक्शन् एनोटेशन टूल् इत्यस्य उपयोगं कुर्वन्तु (DAVID एनोटेशन डाटा धीरेण अद्यतनं भवति अधुना अनुशंसितं नास्ति । तस्य उपयोगः अनुशंसितः अस्तिGo East, सर्वोत्तमम् ऑनलाइन GO संवर्धनविश्लेषणसाधनम्तथाकेवलं एकस्मिन् पदे समृद्धिविश्लेषणं कर्तुं शक्नोति इति एतत् जालपुटं प्रकाशनात् पूर्वं CNS इत्यादिभिः ३५० वारं अधिकं उद्धृतम् अस्ति ।संवर्धनविश्लेषणं कुर्वन्तु, २.एकस्मिन् लेखे GSEA मास्टरं कुर्वन्तु, सुपर विस्तृतं पाठ्यक्रमम्) भिन्नरूपेण अभिव्यक्त जीनानां जीन एनोटेशन (adjusted p-value < 0.05 ) तथा कार्यात्मकसंवर्धनविश्लेषणम्। अस्मिन् दत्तांशसमूहे निम्नलिखितपञ्च दत्तांशवर्गाः सन्ति ।
| नामः | वर्णेतु | दत्तांशसमूहस्य आकारः |
|---|---|---|
| ईसी $ सेट | मस्तिष्कस्य हृदयस्य च अन्तःस्थकोशिकासु सामान्यीकृतजीनअभिव्यक्तिः (३ प्रतिकृतयः) | २०६४४ x ७ |
| ईसी $ जेनेलिस्ट | विभेदितरूपेण अभिव्यक्ताः जीनाः (समायोजितं p-मूल्यं < 0.05) | २०३९ x ७ |
| ईसी$दाविद | DAVID इत्यस्य उपयोगेन विभेदकजीनानां कार्यात्मकसंवर्धनविश्लेषणस्य परिणामाः | १७४ x ५ |
| ईसी $ जीन | जीन तथा logFC | ३७ x २ |
| ईसी $ प्रक्रिया | समृद्धजैविकप्रक्रियाणां कृते चयनितविशेषतासदिशः | 7 |
वयं भिन्नरूपेण अभिव्यक्तजीनानां GO समृद्धमार्गान् द्रष्टुम् इच्छामः, परन्तु चित्रकला आरभ्यतुं पूर्वं अस्माभिः प्रारूपस्य आवश्यकतां पूरयति इति दत्तांशं प्रदातव्यम् ।सामान्यतया आलेखस्य आकर्षणार्थं आवश्यकं दत्तांशं स्वयमेव प्रदत्तं भवति, परन्तु...अस्मिन् संकुले एकं कार्यम् अस्तिcircle_datदत्तांशस्वरूपस्य निवारणे अस्मान् साहाय्यं कर्तुं शक्नोति。circle_datइदं चयनितजीनानां कार्यात्मकसमृद्धिविश्लेषणपरिणामानां तेषां logFC मूल्यानां च संयोजनं कर्तुं शक्नोति, मुख्यतया भिन्नरूपेण अभिव्यक्तजीनानां कृते ।circle_dat उपयोगः अतीव सरलः अस्ति, केवलं द्वयोः दत्तांशयोः पठन्तु । प्रथमे दत्तांशे कार्यात्मकसमृद्धिविश्लेषणपरिणामाः सन्ति, यत्र न्यूनातिन्यूनं चत्वारि स्तम्भाः (कार्यात्मकसमृद्धिविश्लेषणवर्गः, मार्गः, जीनः, समायोजितं p-मूल्यं) सन्ति ।द्वितीयः दत्तांशः चयनितजीनस्य तस्य logFC च अस्ति, एषः दत्तांशः स्रोतः भवितुम् अर्हतिlimmaसांख्यिकीयविश्लेषणस्य परिणामाः (जीवनवृत्तात् टिप्पणी: द्वयोः सञ्चिकायोः अवश्यं ध्यानं ददातुजीनानां नामकरणं कथं भवतिसर्वादिकं सुसंगतं भवतुGene symbol ). उपरि उल्लिखितानि दत्तांशस्वरूपाणि उदाहरणैः सह पश्यामः ।
- #安装已发布的稳定版本
- #install.packages('GOplot')
- #安装github上的开发版本
- #install_github('wencke/wencke.github.io')
- #载入包
- library(GOplot)
- #读入包内自带的数据
- data(EC)
- #查看功能富集分析结果的数据格式
- head(EC$david)
-
- ## Category ID Term
- ## 1 BP GO:0007507 heart development
- ## 2 BP GO:0001944 vasculature development
- ## 3 BP GO:0001568 blood vessel development
- ## 4 BP GO:0048729 tissue morphogenesis
- ## 5 BP GO:0048514 blood vessel morphogenesis
- ## 6 BP GO:0051336 regulation of hydrolase activity
- ## Genes
- ## 1 DLC1, NRP2, NRP1, EDN1, PDLIM3, GJA1, TTN, GJA5, ZIC3, TGFB2, CERKL, GATA6, COL4A3BP, GAB1, SEMA3C, MKL2, SLC22A5, MB, PTPRJ, RXRA, VANGL2, MYH6, TNNT2, HHEX, MURC, MIB1, FOXC2, FOXC1, ADAM19, MYL2, TCAP, EGLN1, SOX9, ITGB1, CHD7, HEXIM1, PKD2, NFATC4, PCSK5, ACTC1, TGFBR2, NF1, HSPG2, SMAD3, TBX1, TNNI3, CSRP3, FOXP1, KCNJ8, PLN, TSC2, ATP6V0A1, TGFBR3, HDAC9
- ## 2 GNA13, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, FOXO1, GJA5, TGFB2, WARS, CERKL, APOE, CXCR4, ANG, SEMA3C, NOS2, MKL2, FGF2, RAPGEF1, PTPRJ, RECK, EFNB2, VASH1, PNPLA6, THY1, MIB1, NUS1, FOXC2, FOXC1, CAV1, CDH2, MEIS1, WT1, CDH5, PTK2, FBXW8, CHD7, PLCD1, PLXND1, FIGF, PPAP2B, MAP2K1, TBX4, TGFBR2, NF1, TBX1, TNNI3, LAMA4, MEOX2, ECSCR, HBEGF, AMOT, TGFBR3, HDAC7
- ## 3 GNA13, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, FOXO1, GJA5, TGFB2, WARS, CERKL, APOE, CXCR4, ANG, SEMA3C, NOS2, MKL2, FGF2, RAPGEF1, PTPRJ, RECK, VASH1, PNPLA6, THY1, MIB1, NUS1, FOXC2, FOXC1, CAV1, CDH2, MEIS1, WT1, CDH5, PTK2, FBXW8, CHD7, PLCD1, PLXND1, FIGF, PPAP2B, MAP2K1, TBX4, TGFBR2, NF1, TBX1, TNNI3, LAMA4, MEOX2, ECSCR, HBEGF, AMOT, TGFBR3, HDAC7
- ## 4 DLC1, ENAH, NRP1, PGF, ZIC2, TGFB2, CD44, ILK, SEMA3C, RET, AR, RXRA, VANGL2, LEF1, TNNT2, HHEX, MIB1, NCOA3, FOXC2, FOXC1, TGFB1I1, WNT5A, COBL, BBS4, FGFR3, TNC, BMPR2, CTNND1, EGLN1, NR3C1, SOX9, TCF7L1, IGF1R, FOXQ1, MACF1, HOXA5, BCL2, PLXND1, CAR2, ACTC1, TBX4, SMAD3, FZD3, SHANK3, FZD6, HOXB4, FREM2, TSC2, ZIC5, TGFBR3, APAF1
- ## 5 GNA13, CAV1, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, CDH2, MEIS1, WT1, TGFB2, WARS, PTK2, CERKL, APOE, CXCR4, ANG, SEMA3C, PLCD1, NOS2, MKL2, PLXND1, FIGF, FGF2, PTPRJ, TGFBR2, TBX4, NF1, TBX1, TNNI3, PNPLA6, VASH1, THY1, NUS1, MEOX2, ECSCR, AMOT, HBEGF, FOXC2, FOXC1, HDAC7
- ## 6 CAV1, XIAP, AGFG1, ADORA2A, TNNC1, TBC1D9, LEPR, ABHD5, EDN1, ASAP2, ASAP3, SMAP1, TBC1D12, ANG, TBC1D14, MTCH1, TBC1D13, TBC1D4, TBC1D30, DHCR24, HIP1, VAV3, NOS1, NF1, MYH6, RICTOR, TBC1D22A, THY1, PLCE1, RNF7, NDEL1, CHML, IFT57, ACAP2, TSC2, ERN1, APAF1, ARAP3, ARAP2, ARAP1, HTR2A, F2R
- ## adj_pval
- ## 1 0.000002170
- ## 2 0.000010400
- ## 3 0.000007620
- ## 4 0.000119000
- ## 5 0.000720000
- ## 6 0.001171166
-
- #查看基因的数据格式
- head(EC$genelist)
-
- ## ID logFC AveExpr t P.Value adj.P.Val B
- ## 1 Slco1a4 6.645388 1.2168670 88.65515 1.32e-18 2.73e-14 29.02715
- ## 2 Slc19a3 6.281525 1.1600468 69.95094 2.41e-17 2.49e-13 27.62917
- ## 3 Ddc 4.483338 0.8365231 65.57836 5.31e-17 3.65e-13 27.18476
- ## 4 Slco1c1 6.469384 1.3558865 59.87613 1.62e-16 8.34e-13 26.51242
- ## 5 Sema3c 5.515630 2.3252117 58.53141 2.14e-16 8.81e-13 26.33626
- ## 6 Slc38a3 4.761755 0.9218670 54.11559 5.58e-16 1.76e-12 25.70308
इनपुट् दत्तांशस्वरूपद्वयं अवगत्य भवान् उपयोक्तुं शक्नोतिcirlce_datरेखाचित्रदत्तांशं जनयितुं कार्यम् ।
- # 生成画图所需的数据格式
- circ <- circle_dat(EC$david, EC$genelist)
-
- head(circ)
-
- ## category ID term count genes logFC adj_pval
- ## 1 BP GO:0007507 heart development 54 DLC1 -0.9707875 2.17e-06
- ## 2 BP GO:0007507 heart development 54 NRP2 -1.5153173 2.17e-06
- ## 3 BP GO:0007507 heart development 54 NRP1 -1.1412315 2.17e-06
- ## 4 BP GO:0007507 heart development 54 EDN1 1.3813006 2.17e-06
- ## 5 BP GO:0007507 heart development 54 PDLIM3 -0.8876939 2.17e-06
- ## 6 BP GO:0007507 heart development 54 GJA1 -0.8179480 2.17e-06
- ## zscore
- ## 1 -0.8164966
- ## 2 -0.8164966
- ## 3 -0.8164966
- ## 4 -0.8164966
- ## 5 -0.8164966
- ## 6 -0.8164966
circवस्तुनः अष्टस्तम्भाः दत्तांशस्तम्भाः सन्ति, यथा
category: बीपी (जैविक प्रक्रिया), सीसी (कोशिकीय घटक) या एमएफ (आणविक कार्य)
ID: GO id (वैकल्पिकस्तम्भः, यदि भवान् GO id इत्यस्य आधारेण न भवति इति कार्यात्मकविश्लेषणसाधनस्य उपयोगं कर्तुम् इच्छति तर्हि ID स्तम्भं चयनं कर्तुं न शक्नोति; अत्र ID KEGG ID अपि भवितुम् अर्हति)
term: GO मार्ग
count: प्रत्येकस्मिन् मार्गे जीनानां संख्या
जीन: जीननाम - logFC: प्रत्येकस्य जीनस्य logFC मूल्यम्
adj_pval: समायोजितं p मूल्यं, adj_pval<0.05 युक्ताः मार्गाः महत्त्वपूर्णतया समृद्धाः इति मन्यन्ते
zscore: zscore सांख्यिकीयसामान्यीकरणपद्धतिं न निर्दिशति, परन्तु जैविकप्रक्रियायाः (/आणविककार्यं/कोशिकीयघटकस्य) न्यूनतायाः (नकारात्मकमूल्यं) अथवा वर्धनस्य (सकारात्मकमूल्यं) अधिकसंभावना अस्ति वा इति अनुमानं कर्तुं सुलभतया गणितं मूल्यम् अस्तिगणनाविधिः प्रत्येकं मार्गे जीनसङ्ख्यायाः वर्गमूलेन विभक्तं अप-रेगुलेटेड् जीनानां संख्यां न्यूनीकृत्य डाउन-रेगुलेटेड् जीनानां संख्यां भवति
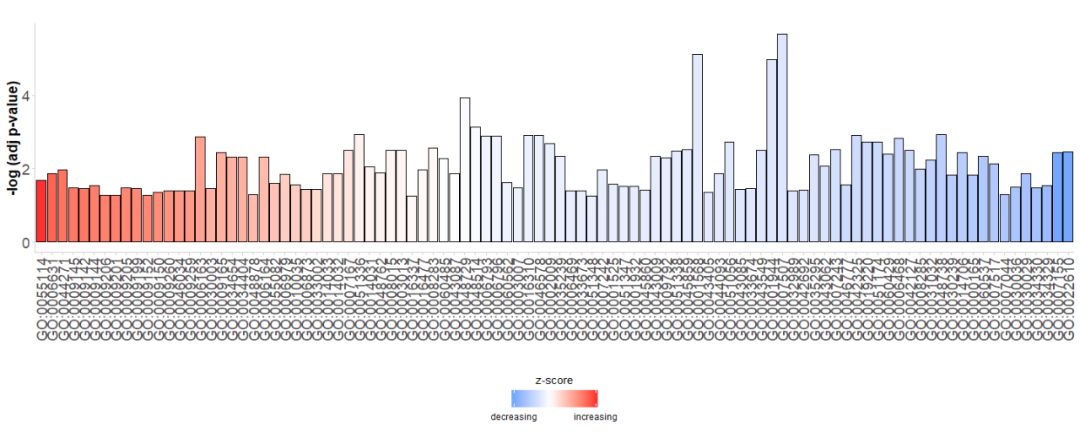
यदा वयं प्रथमवारं दत्तांशं पश्यामः तदा वयं ग्राफ् तः यथासंभवं मार्गं दर्शयितुम् इच्छामः, अपि च बहुमूल्यमार्गान् अन्वेष्टुम् इच्छामः, अतः महत्त्वस्य मूल्याङ्कनार्थं केचन मापदण्डाः आवश्यकाः प्रायः नमूनादत्तांशस्य वर्णनार्थं बार चार्ट्स् इत्यस्य उपयोगः भवति, अतः वयं शीघ्रं सुन्दरं बार चार्ट् निर्मातुं GOBar फंक्शन् इत्यस्य उपयोगं कर्तुं शक्नुमः ।
प्रथमं प्रत्यक्षतया सरलं बार चार्ट् उत्पद्यतेGO Terms, तेषां मतेzscoreदण्डान् क्रमयन्तु;-log(adj p-value);वर्णः प्रतिनिधियतिzscore, नीलवर्णः सूचयतिz-scoreनकारात्मकं मूल्यं भवति, तत्सम्बद्धमार्गे जीनव्यञ्जनस्य न्यूनतायाः अधिका सम्भावना भवति, रक्तवर्णेन सूचितम्z-score सकारात्मकं मूल्यं भवति, तत्सम्बद्धमार्गे जीनव्यञ्जनस्य वृद्धिः अधिका भवति । यदि इष्टं भवति तर्हि order.by.zscore इति पैरामीटर् FALSE इति सेट् कृत्वा क्रमं परिवर्तयितुं शक्यते, अस्मिन् सति पट्टिकाः तेषां महत्त्वस्य आधारेण क्रमिताः भवन्ति ।
- # 生成简单的条形图
- GOBar(subset(circ, category == 'BP'))
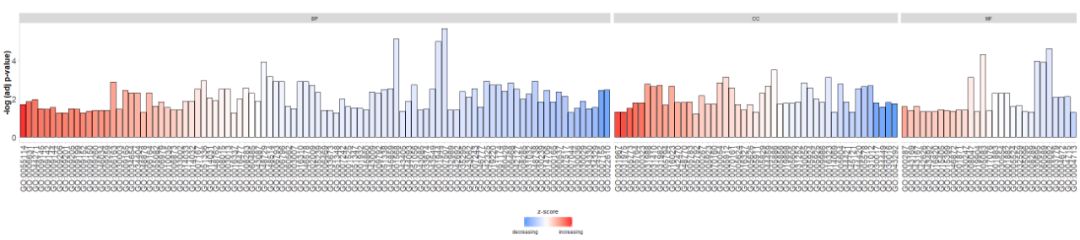
#GOBar(subset(circ, category == 'BP',order.by.zscore=FALSE))तदतिरिक्तं, चैनलस्य वर्गानुसारं बार चार्ट् आकर्षयितुं display पैरामीटर् परिवर्तयन्तु ।
- #根据通路的类别来绘制条形图
- GOBar(circ, display = 'multiple')
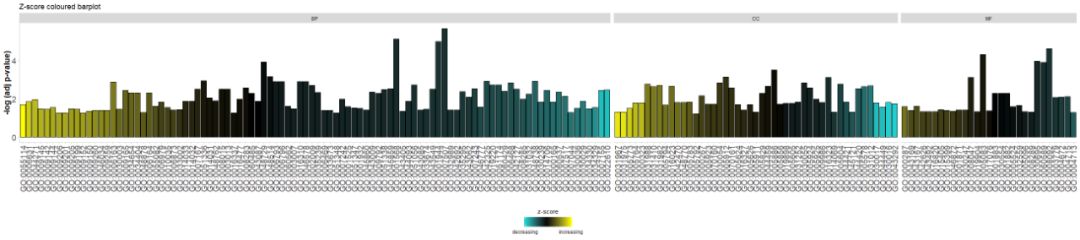
शीर्षकं योजयित्वा पैरामीटर्स् इत्यस्य उपयोगं कुर्वन्तुzsc.colपरिवर्तयzscores वर्णः ।
- # Facet the barplot, add a title and change the colour scale for the z-score
- GOBar(circ, display = 'multiple', title = 'Z-score coloured barplot', zsc.col = c('yellow', 'black', 'cyan'))
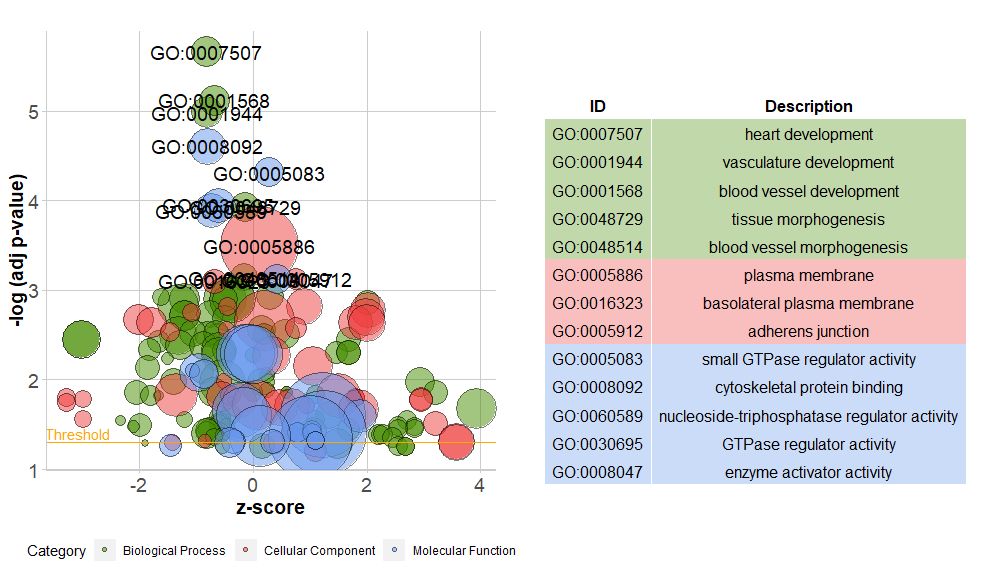
बार चार्ट्स् अतीव सामान्याः सुलभाः च सन्ति, परन्तु वयं दत्तांशस्य विषये अधिकानि सूचनानि प्रदर्शयितुं बबल चार्ट्स् इत्यस्य उपयोगं कर्तुं शक्नुमः ।
क्षैतिजः अक्षः अस्तिzscore;लम्ब-अक्षः अस्ति-log(adj p-value), बारचार्टस्य सदृशं यत्किमपि अधिकं भवति तथा तथा वृत्तस्य क्षेत्रफलं तत्सम्बद्धमार्गे जीनसङ्ख्यायाः सह सम्बद्धं भवति;circ$count );द्वारा प्रविष्टुं शक्यते?GOBubble चित्रस्य सर्वाणि मापदण्डानि परिवर्तयितुं GOBubble फंक्शन् इत्यस्य सहायतापृष्ठं पश्यन्तु । पूर्वनिर्धारितरूपेण प्रत्येकं वृत्तं तत्सम्बद्धेन GO ID इत्यनेन चिह्नितं भवति, तथा च GO ID तथा GO पदयोः तत्सम्बद्धं सम्बन्धं दर्शयति सारणी अपि दक्षिणभागे प्रदर्शिता भवतिपैरामीटर् सेट् कर्तुं शक्यतेtable.legendकृतेFALSE तत् गोपयितुं । यदि भवान् मार्गविवरणं प्रदर्शयितुम् इच्छति तर्हि पैरामीटर् ID FALSE इति सेट् कुर्वन्तु ।परन्तु सीमितस्थानस्य, आच्छादितवृत्तानां च कारणात् सर्वाणि वृत्तानि चिह्नितानि न भवन्ति, केवलं...-log(adj p-value) > 3(पूर्वनिर्धारितं ५ अस्ति)।
- # 生成泡泡图,并展示-log(adj p-value) > 3 的通路的GO ID
- GOBubble(circ, labels = 3)
यदि भवान् बबलचार्ट् मध्ये शीर्षकं योजयितुम् इच्छति, अथवा वृत्तस्य वर्णं निर्दिशति तथा च प्रत्येकस्य वर्गस्य मार्गं पृथक् प्रदर्शयितुम् इच्छति तथा च प्रदर्शितं GO ID सीमां परिवर्तयितुं इच्छति तर्हि भवान् निम्नलिखितमापदण्डान् योजयितुं शक्नोति:
GOBubble(circ, title = 'Bubble plot', colour = c('orange', 'darkred', 'gold'), display = 'multiple', labels = 3)
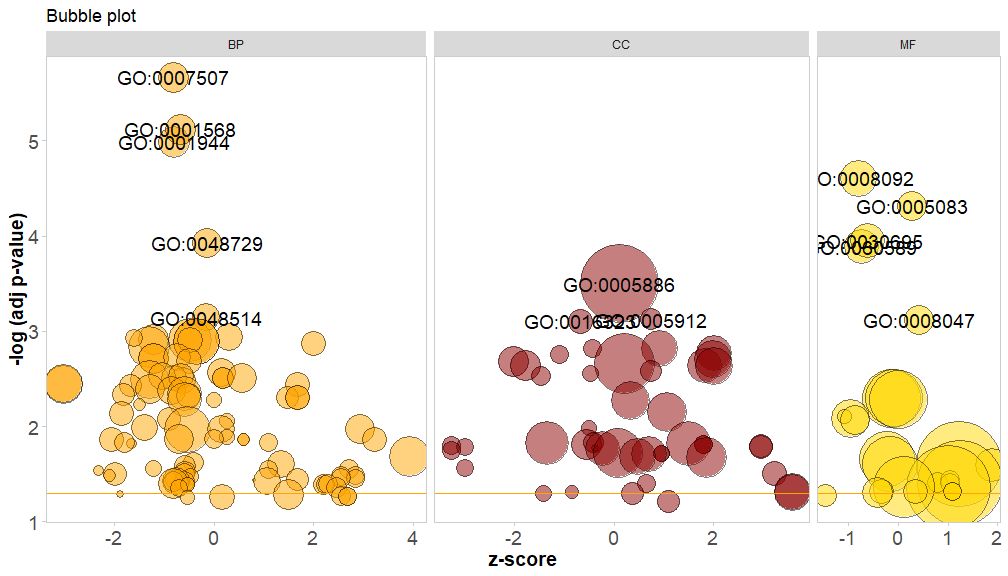
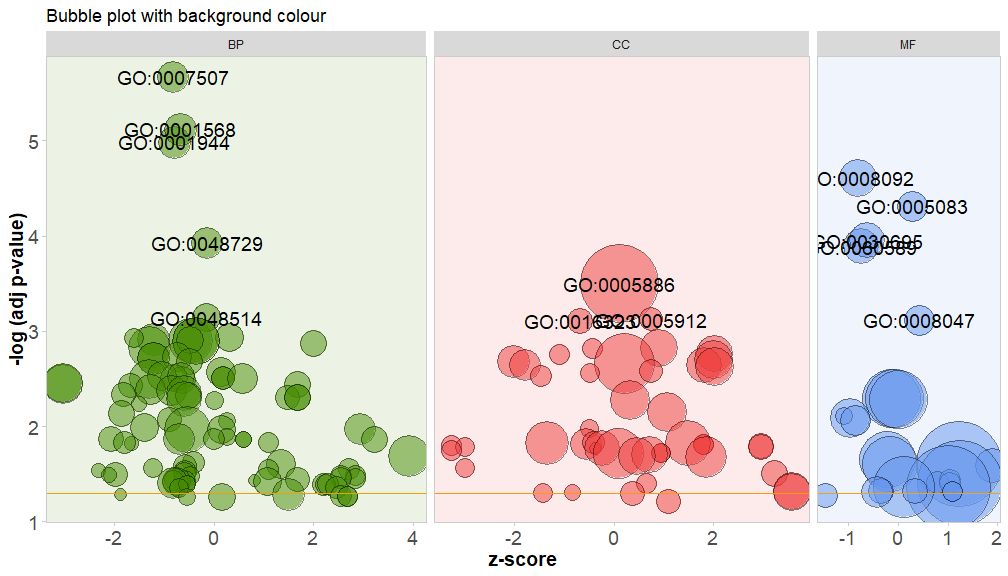
bg.col इति पैरामीटर् TRUE इति सेट् कृत्वा चैनल् इत्यस्य वर्गस्य पृष्ठभूमिं रङ्गयन्तु ।
GOBubble(circ, title = 'Bubble plot with background colour', display = 'multiple', bg.col = T, labels = 3)
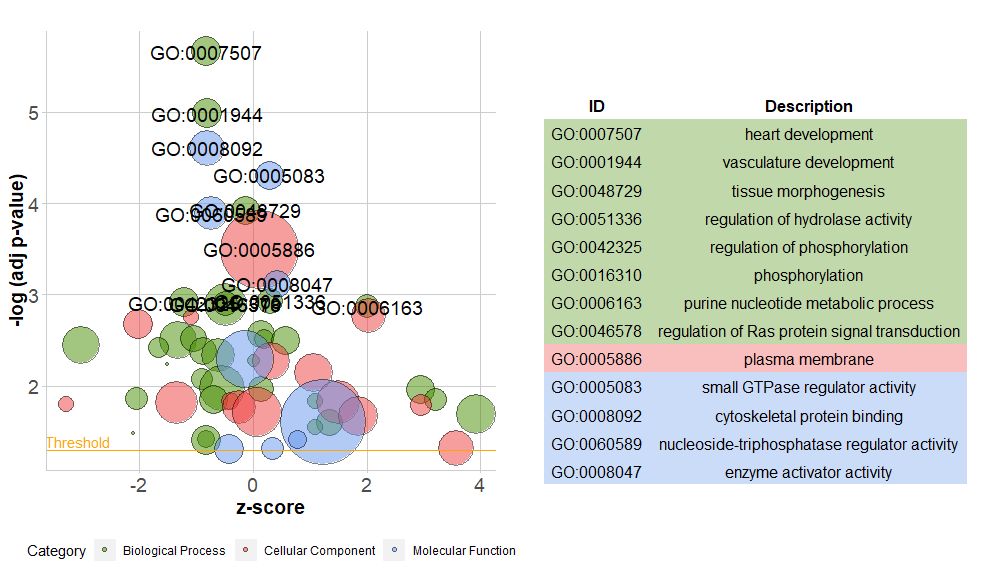
संकुलस्य नूतनसंस्करणे नूतनं कार्यं भवतिreduce_overlap . मार्गाः GO इत्यत्र । अनावश्यकपदानां संख्यां न्यूनीकृत्य कथानकानाम् (बबलप्लॉट् इत्यादीनां) पठनीयतायां महत्त्वपूर्णं सुधारः भवति ।
- # reduce_overlap,参数设置为0.75
- reduced_circ <- reduce_overlap(circ, overlap = 0.75)
-
- GOBubble(reduced_circ, labels = 2.8)
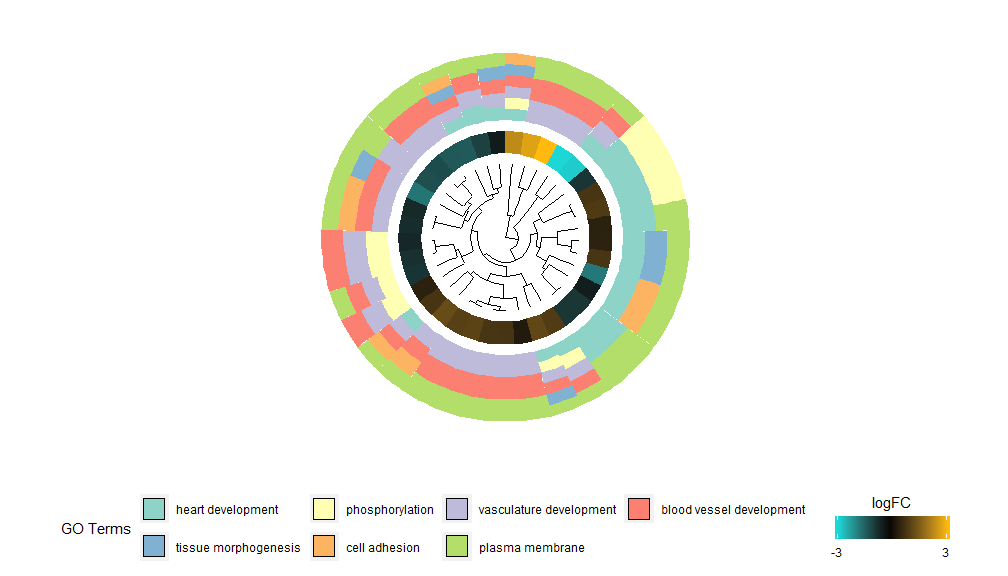
यद्यपि सर्वाणि सूचनानि दर्शयन् आलेखः अस्मान् अन्वेष्टुं साहाय्यं कर्तुं शक्नोति यत् के मार्गाः अधिकतया सार्थकाः सन्ति, तथापि वास्तविकता अद्यापि भवन्तः दत्तांशैः सह पुष्टिं कर्तुम् इच्छन्ति इति परिकल्पनाविचारयोः उपरि निर्भरं भवति, महत्त्वपूर्णाः मार्गाः च भवतः रुचिः न भवेयुः इति अनिवार्यम् अतः मार्गसमूहस्य बहुमूल्यं चयनं हस्तचलितरूपेण (EC$process ), अस्माकं कृते अस्य विशिष्टस्य मार्गसमूहस्य विषये अधिकविस्तृतसूचनाः दर्शयितुं चित्रस्य आवश्यकता वर्तते ।परन्तु एतानि आकृतयः प्रस्तुत्य एकः समस्या उत्पद्यते यत् कदाचित् तस्य व्याख्या कठिना भवतिzscore सूचना प्रदत्ता।किन्तु, एषा गणनाविधिः सार्वत्रिकः नास्ति यथा उपरि दर्शितं, एषा केवलं अप-रेगुलेटेड् जीनानां संख्यां न्यूनीकृत्य प्रत्येकस्मिन् मार्गे जीनसङ्ख्यायाः वर्गमूलेन विभक्तं भवतिGOCircleपरिणामी आलेखः अपि एतत् तथ्यं बोधयति ।
वृत्तचित्रस्य बाह्यवृत्तं प्रत्येकस्य मार्गस्य जीनानां logFC मूल्यं विकीर्णबिन्दुरूपेण दर्शयति । रक्तवृत्तानि अपरेग्युलेशनं सूचयन्ति नीलवर्णं च अधः रेगुलेशनं सूचयन्ति ।पैरामीटर्स् उपयोक्तुं शक्यन्तेlfc.col वर्णं परिवर्तयन्तु। एतेन अपि व्याख्यायते यत् केषुचित् सन्दर्भेषु अत्यन्तं महत्त्वपूर्णमार्गेषु शून्यस्य समीपे zscores किमर्थं भवति । शून्यस्य zscore इत्यस्य अर्थः न भवति यत् चैनलः अमहत्त्वपूर्णः अस्ति । इदं केवलं दर्शयति यत् zscore एकः रूक्षः मापः अस्ति, यतः स्पष्टतया zscore जैविकप्रक्रियासु व्यक्तिगतजीनानां कार्यात्मकस्तरं सक्रियीकरणनिर्भरतां च न गृह्णाति
GOCircle(circ)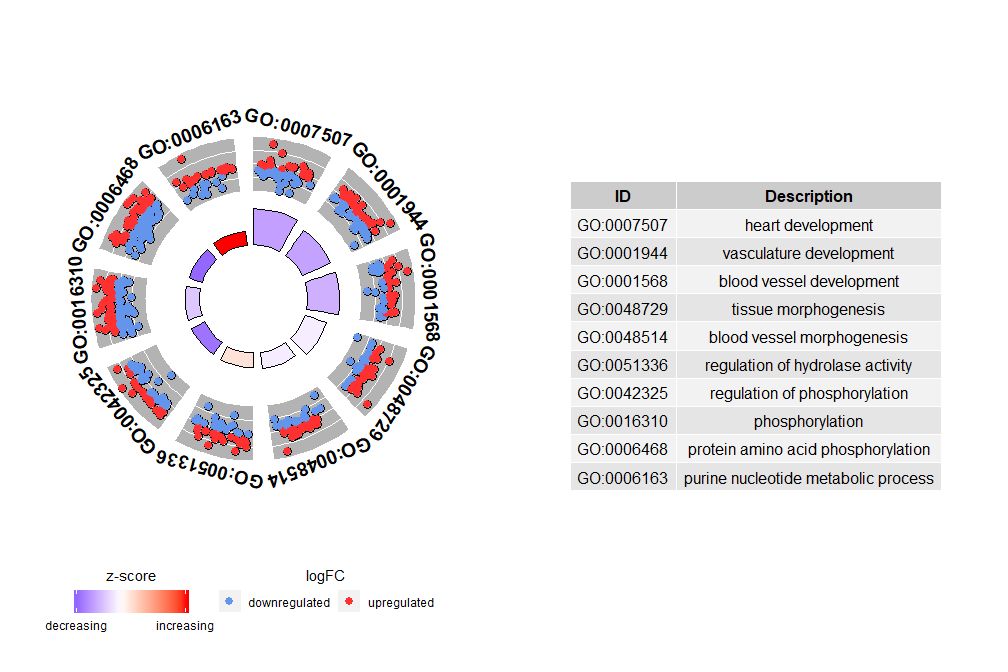
nsub पैरामीटर्स् संख्याः अथवा वर्णसदिशः सेट् कर्तुं शक्यन्ते । यदि वर्णसदिशः अस्ति तर्हि तस्मिन् GO ID अथवा प्रदर्शयितुं मार्गः अस्ति;
- # 生成特定通路的圈图
- IDs <- c('GO:0007507', 'GO:0001568', 'GO:0001944', 'GO:0048729', 'GO:0048514', 'GO:0005886', 'GO:0008092', 'GO:0008047')
- GOCircle(circ, nsub = IDs)
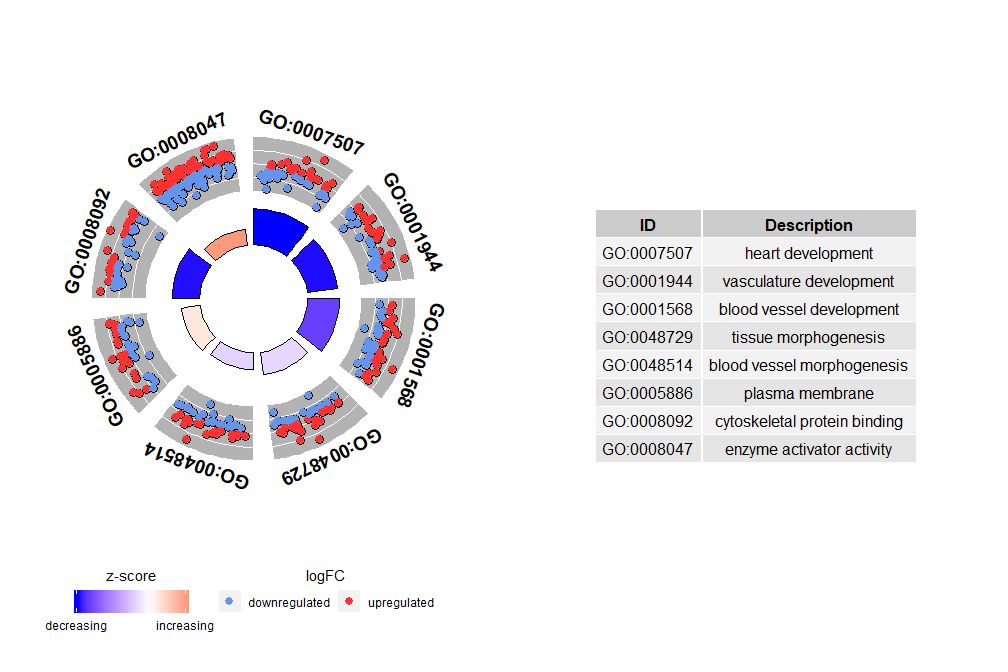
यदि nsub संख्यात्मकः सदिशः अस्ति तर्हि संख्या प्रदर्शयितुं संख्यां परिभाषयति । इदं इनपुट् डाटाफ्रेम् इत्यस्य प्रथमपङ्क्तौ आरभ्यते । एतत् दृश्यीकरणं केवलं लघुदत्तांशैः सह कार्यं करोति । अधिकतमं चैनलानां संख्या पूर्वनिर्धारितरूपेण १२ भवति । यद्यपि चैनलानां संख्या न्यूनीभवति तथापि प्रदर्शितसूचनायाः परिमाणं वर्धते ।
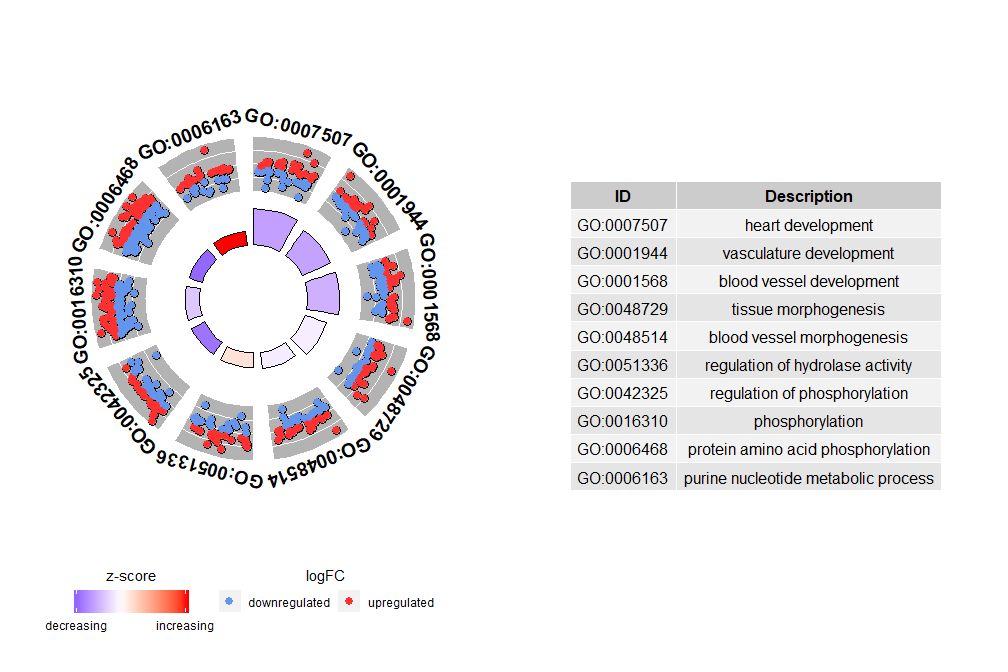
- # 圈图展示数据前十个通路
- GOCircle(circ, nsub = 10)
GOChord चयनितजीनानां मार्गानाञ्च सम्बन्धं जीनानां logFC च प्रदर्शयितुं शक्नोति ।प्रथमं भवद्भिः एकं matrix input करणीयम्, यत् भवन्तः स्वयमेव निर्मातुं शक्नुवन्ति0-1Matrix, भवन्तः फंक्शन्स् अपि उपयोक्तुं शक्नुवन्तिchord_dat निर्माणं कुरुत। अस्य कार्यस्य त्रयः मापदण्डाः सन्ति : दत्तांशः, जीनः, प्रक्रिया च, येषु अन्तिमयोः मापदण्डयोः न्यूनातिन्यूनं एकः मापदण्डः भवितुमर्हति ।अथ कार्यम्circle_datकार्यात्मकविश्लेषणानाम् परिणामैः सह अभिव्यक्तिदत्तांशं संयोजयन्तु।
बार चार्ट्स् तथा बबल चार्ट्स् भवन्तं दत्तांशस्य प्रथमानुभूतिम् दातुं शक्नुवन्ति अधुना, भवान् केचन जीनाः मार्गाः च चयनं कर्तुं शक्नुवन्ति ये वयं मूल्यवान् इति मन्यामहे यद्यपि GOCircle मार्गेषु जीनानां अभिव्यक्तिमूल्यं प्रदर्शयितुं एकं स्तरं योजयति, तथापि अस्मिन् एकस्या सूचनायाः अभावः अस्ति जीनानां बहुमार्गाणां च सम्बन्धेषु। केचन जीनाः बहुविधप्रक्रियाभिः सह सम्बद्धाः सन्ति वा इति चिन्तयितुं न सुकरम् । GOChord GOCircle इत्यस्य दोषाणां पूर्तिं करोति। उत्पन्नदत्तांशस्य पङ्क्तयः जीनाः सन्ति तथा च स्तम्भाः मार्गाः सन्ति "0" इत्यस्य अर्थः अस्ति यत् जीनः मार्गे न नियुक्तः, "1" च विपरीतम् ।
- # 找到感兴趣的的基因,这里我们以EC$genes为例
- head(EC$genes)
-
- ## ID logFC
- ## 1 PTK2 -0.6527904
- ## 2 GNA13 0.3711599
- ## 3 LEPR 2.6539788
- ## 4 APOE 0.8698346
- ## 5 CXCR4 -2.5647537
- ## 6 RECK 3.6926860
-
- # 获得感兴趣基因的通路
- EC$process
-
- ## [1] "heart development" "phosphorylation"
- ## [3] "vasculature development" "blood vessel development"
- ## [5] "tissue morphogenesis" "cell adhesion"
- ## [7] "plasma membrane"
-
- # 使用chord_dat构建矩阵
- chord <- chord_dat(circ, EC$genes, EC$process)
- head(chord)
-
- ## heart development phosphorylation vasculature development
- ## PTK2 0 1 1
- ## GNA13 0 0 1
- ## LEPR 0 0 1
- ## APOE 0 0 1
- ## CXCR4 0 0 1
- ## RECK 0 0 1
- ## blood vessel development tissue morphogenesis cell adhesion
- ## PTK2 1 0 0
- ## GNA13 1 0 0
- ## LEPR 1 0 0
- ## APOE 1 0 0
- ## CXCR4 1 0 0
- ## RECK 1 0 0
- ## plasma membrane logFC
- ## PTK2 1 -0.6527904
- ## GNA13 1 0.3711599
- ## LEPR 1 2.6539788
- ## APOE 1 0.8698346
- ## CXCR4 1 -2.5647537
- ## RECK 1 3.6926860
उदाहरणे वयं द्वौ मापदण्डौ पारितवन्तः यदि केवलं जीनमापदण्डः निर्दिष्टः अस्ति तर्हि परिणामः चयनितजीनानां सूचीः सर्वेषां प्रक्रियानिर्माणानां च न्यूनातिन्यूनं एकेन निर्दिष्टेन जीनेन सह भवति ।0-1matrix यदि केवलं निर्दिष्टं भवतिprocessपैरामीटर्स्, परिणामः अस्ति यत् सर्वे जीनाः उत्पद्यन्ते0-1 सूचीयां न्यूनातिन्यूनं एकस्याः प्रक्रियायाः नियुक्तानां जीनानां आकृतिः । अवगच्छन्तु यत् केवलं जीनानि प्रक्रियामापदण्डानि च निर्दिश्य अत्यन्तं विशालं 0-1 आकृतिः भवितुम् अर्हति, यस्य परिणामेण दृश्यीकरणस्य परिणामाः भ्रमिताः भवितुम् अर्हन्ति ।
- head(circ)
-
- ## category ID term count genes logFC adj_pval
- ## 1 BP GO:0007507 heart development 54 DLC1 -0.9707875 2.17e-06
- ## 2 BP GO:0007507 heart development 54 NRP2 -1.5153173 2.17e-06
- ## 3 BP GO:0007507 heart development 54 NRP1 -1.1412315 2.17e-06
- ## 4 BP GO:0007507 heart development 54 EDN1 1.3813006 2.17e-06
- ## 5 BP GO:0007507 heart development 54 PDLIM3 -0.8876939 2.17e-06
- ## 6 BP GO:0007507 heart development 54 GJA1 -0.8179480 2.17e-06
- ## zscore
- ## 1 -0.8164966
- ## 2 -0.8164966
- ## 3 -0.8164966
- ## 4 -0.8164966
- ## 5 -0.8164966
- ## 6 -0.8164966
-
- # Generate the matrix with a list of selected genes
- chord_genes <- chord_dat(data = circ, genes = EC$genes)
- head(chord_genes)
-
- ## heart development vasculature development blood vessel development
- ## PTK2 0 1 1
- ## GNA13 0 1 1
- ## LEPR 0 1 1
- ## APOE 0 1 1
- ## CXCR4 0 1 1
अयं चार्टः उच्च-आयामी-दत्तांशस्य लघुतरं उपसमूहं दर्शयितुं उद्दिष्टः अस्ति । मुख्यतया द्वौ मापदण्डौ स्तः येषां समायोजनं कर्तुं शक्यते ।gene.orderतथाnlfc . जीन्स् पैरामीटर् 'logFC', 'alphabetical', 'none' इति निर्दिष्टुं शक्यते । वस्तुतः, वयं सामान्यतया जीन्स् पैरामीटर् logFC इति निर्दिशन्ति nlfc पैरामीटर् अस्य कार्यस्य महत्त्वपूर्णेषु पैरामीटर् मध्ये एकः अस्ति, यतः एतत् प्रत्येकं जीनस्य 0 वा अधिकानि logFC मूल्यानि कथं मैट्रिक्समध्ये प्रस्तुतानि सन्ति इति नियन्त्रयितुं शक्नोति; अतः दोषाणां परिहाराय अस्माभिः पैरामीटर्स् निर्दिष्टव्याः ।
यथा, यदि भवतां समीपे logFC मूल्यानि विना मैट्रिक्सः अस्ति तर्हि भवता सेट् कर्तव्यम्nlfc=0 ; पूर्वनिर्धारितं मूल्यं "1" अस्ति यतोहि अधिकांशकालं प्रतिजीनं केवलं एकं logFC मूल्यं भविष्यति इति विश्वासः अस्ति । logFC इत्यस्य प्रतिनिधित्वं कुर्वतां वर्णयुक्तानां आयतानां मध्ये स्थानं परिभाषितुं space पैरामीटर् इत्यस्य उपयोगं कुर्वन्तु । gene.size पैरामीटर् जीननामस्य फॉन्ट् आकारं निर्दिशति, gene.space च जीननामानां मध्ये स्पेस आकारं निर्दिशति ।
- chord <- chord_dat(data = circ, genes = EC$genes, process = EC$process)
- GOChord(chord, space = 0.02, gene.order = 'logFC', gene.space = 0.25, gene.size = 5)
-
- ## Warning: Using size for a discrete variable is not advised.
-
- ## Warning: Removed 7 rows containing missing values (geom_point).
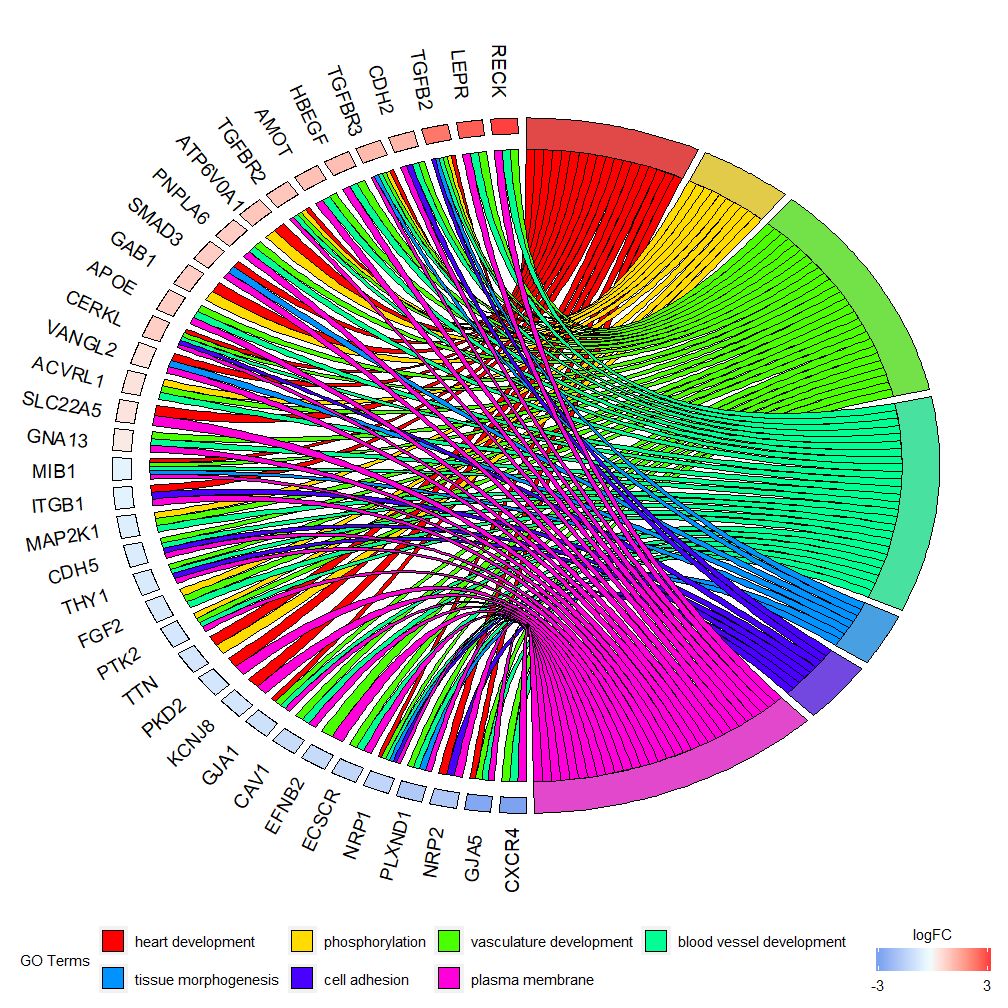
logFC मूल्यानुसारं सेट् कर्तुं शक्यतेgene.order=‘logFC’ , जीनानि तेषां logFC मूल्यानुसारं क्रमबद्धानि भवन्ति । कदाचित् चित्रं किञ्चित् जनसङ्ख्यायुक्तं भवितुम् अर्हति तथा च दर्शितानां जीनानां वा मार्गानाम् संख्यां न्यूनीकर्तुं सीमामापदण्डस्य उपयोगेन एतत् स्वचालितं कर्तुं शक्यते । सीमा द्वौ कटऑफ-मूल्यौ युक्तः सदिशः अस्ति (पूर्वनिर्धारितं c(0,0) अस्ति) । प्रथमं मूल्यं न्यूनतमं मार्गसङ्ख्यां निर्दिशति येषु जीनस्य नियुक्तिः भवितुमर्हति । द्वितीयं मूल्यं मार्गे नियुक्तानां जीनानां संख्यां निर्धारयति ।
- # 仅显示分配给至少三个通路的基因
- GOChord(chord, limit = c(3, 0), gene.order = 'logFC')
-
- ## Warning: Using size for a discrete variable is not advised.
-
- ## Warning: Removed 7 rows containing missing values (geom_point).
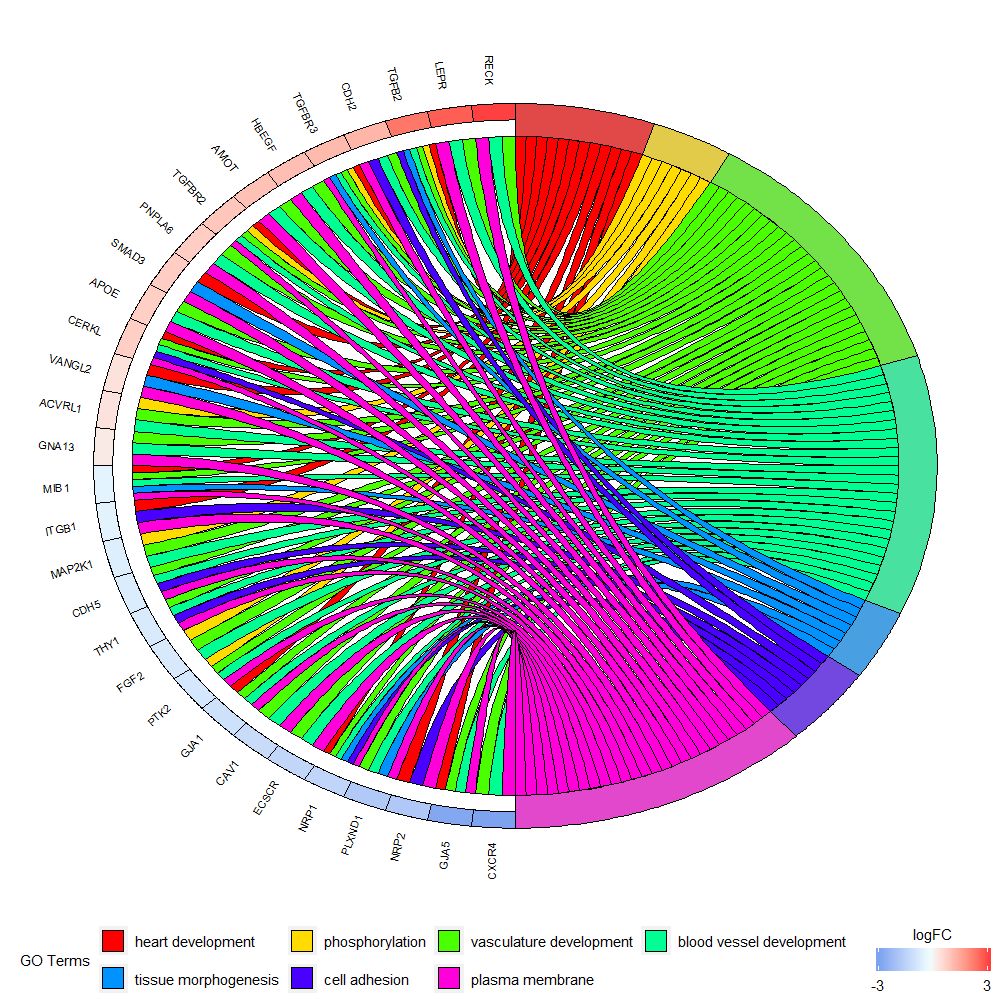
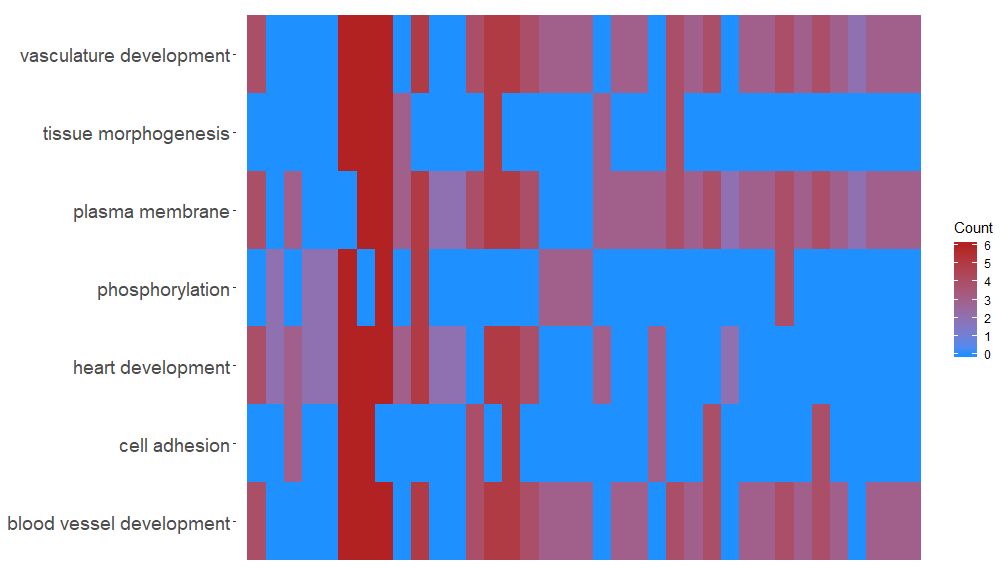
GOHeat कार्यं GOChord इत्यस्य सदृशं तापनक्शस्य उपयोगेन जीनानां मार्गाणां च सम्बन्धं प्रदर्शयितुं शक्नोति । जैविकप्रक्रियाः क्षैतिजरूपेण प्रदर्शिताः भवन्ति, जीनाः लम्बवत् प्रदर्शिताः भवन्ति । प्रत्येकं स्तम्भं लघुआयतेषु विभक्तं भवति, तथा च वर्णः सामान्यतया logFC मूल्यस्य उपरि निर्भरं भवति । तदतिरिक्तं समानकार्यमार्गैः समृद्धाः जीनाः समूहीकृताः आसन् । nlfc पैरामीटर्स् इत्यस्य आधारेण हीट्मैप् वर्णचयनार्थं द्वौ मोडौ स्तः । यदि nlfc = 0 भवति तर्हि वर्णः प्रत्येकस्य जीनस्य समृद्धमार्गसङ्ख्या भवति । विस्तरेण उदाहरणानि पश्यन्तु : १.
- # First, we use the chord object without logFC column to create the heatmap
- GOHeat(chord[,-8], nlfc = 0)
GOHeat(chord[,-8])वर्णः nlfc = 1 इति सन्दर्भे जीनस्य logFC इत्यनेन सह सङ्गच्छते
GOHeat(chord, nlfc = 1, fill.col = c('red', 'yellow', 'green'))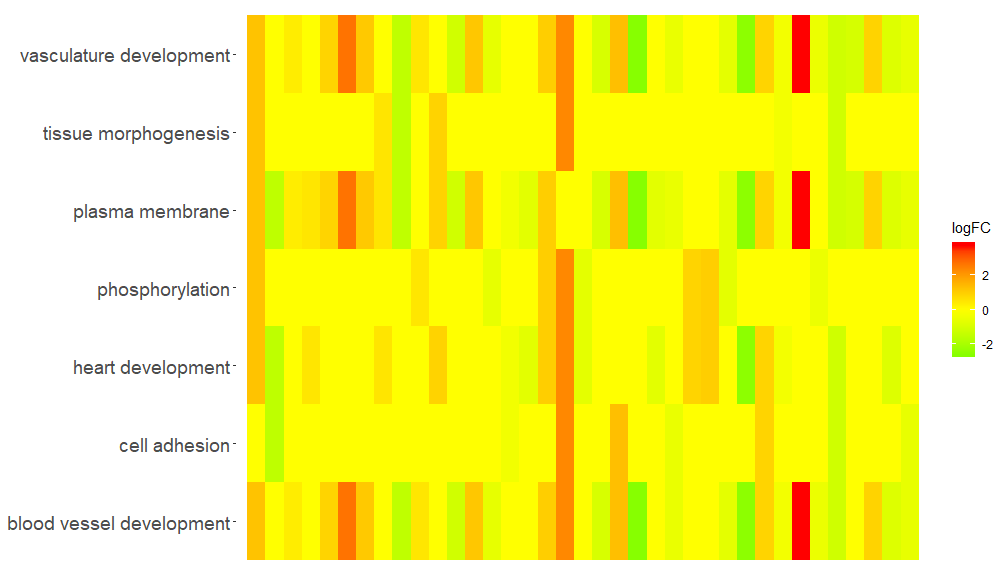
GOCluster कार्यक्षमतायाः पृष्ठतः विचारः यथासम्भवं सूचनां प्रदर्शयितुं भवति । अत्र उदाहरणम् अस्ति : १.
- GOCluster(circ, EC$process, clust.by = 'logFC', term.width = 2)
-
- ## Warning: Using size for a discrete variable is not advised.
-
- ## Warning: Removed 7 rows containing missing values (geom_point).
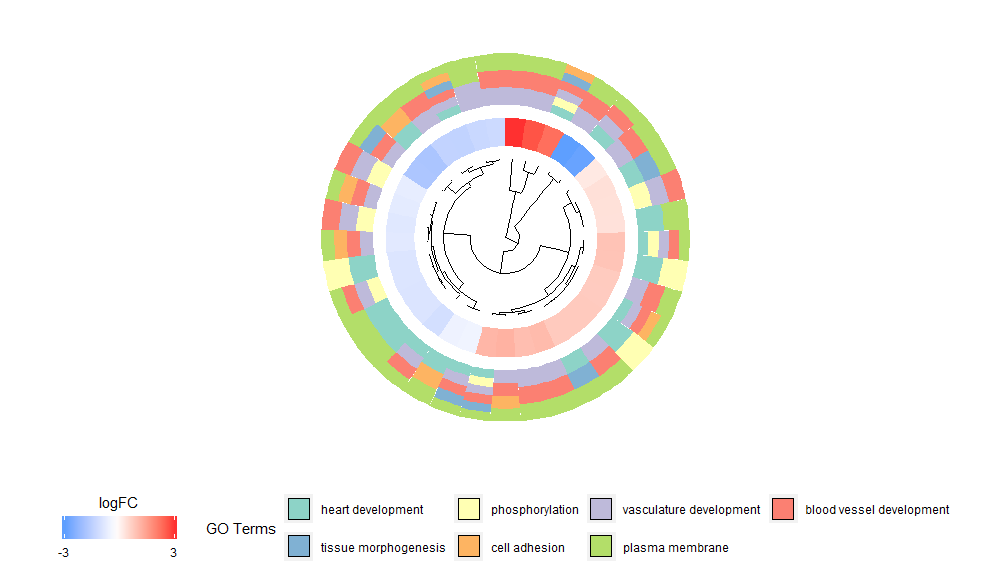
श्रेणीबद्धसमूहीकरणं जीनव्यञ्जनस्य कृते एकः लोकप्रियः अनिरीक्षितसमूहविश्लेषणपद्धतिः अस्ति या अभिव्यक्तिप्रतिमानेन जीनानां एकत्र निष्पक्षसमूहीकरणं सुनिश्चितं करोति, येन समूहेषु सह-नियन्त्रितानां अथवा कार्यात्मकरूपेण सम्बद्धानां जीनानां बहुसमूहाः भवितुं शक्नुवन्ति GOCluster इत्यस्य उपयोगः भवतिhclust विधि जीनव्यञ्जनप्रोफाइलस्य श्रेणीबद्धसमूहीकरणं करोति । यदि भवान् दूरी मेट्रिकं अथवा क्लस्टरिंग् एल्गोरिदम् परिवर्तयितुम् इच्छति तर्हि क्रमशः पैरामीटर् मेट्रिक् तथा क्लस्ट् इत्येतयोः उपयोगं कुर्वन्तु परिणामी डेन्ड्रोग्रामं ggdendro इत्यस्य साहाय्येन परिवर्तयितुं ggplot2 इत्यनेन सह दृश्यमानं कर्तुं च शक्यते । वृत्तविन्यासं चिनुत यतः एतत् न केवलं प्रभावी अपितु दृग्गतरूपेण अपि आकर्षकम् अस्ति। डेण्ड्रोग्रामस्य पार्श्वे प्रथमं वृत्तं जीनस्य logFC इत्यस्य प्रतिनिधित्वं करोति, यत् वस्तुतः समूहवृक्षस्य पत्रम् अस्ति । यदि भवान् बहुविपरीततासु रुचिं लभते तर्हि भवान् nlfc पैरामीटर् परिवर्तयितुं शक्नोति, पूर्वनिर्धारितरूपेण "1" इति सेट् भवति अतः केवलं एकं रिंग् आकृष्टं भवति । logFC मूल्यानि उपयोक्तृ-निर्धार्य-वर्ण-मापदण्डस्य (lfc.col) उपयोगेन वर्ण-सङ्केताः भवन्ति; उत्तमं दृश्यमानं भवितुं चैनलानां संख्या न्यूनीकृता अस्ति, तथा च term.col इति पैरामीटर् इत्यस्य उपयोगेन चैनलानां वर्णं परिवर्तयितुं शक्यते ।अद्यापि उपलब्धम् अस्ति?GOCluster पैरामीटर्स् कथं परिवर्तयितव्याः इति द्रष्टुं । अस्य कार्यस्य महत्त्वपूर्णः पैरामीटर् cluster.by अस्ति, यत् जीनव्यञ्जनप्रतिमानेन ('logFC', यथा उपरि दर्शितम्) अथवा कार्यात्मकवर्गैः ('terms') द्वारा क्लस्टरं कर्तुं निर्दिष्टुं शक्यते
- GOCluster(circ, EC$process, clust.by = 'term', lfc.col = c('darkgoldenrod1', 'black', 'cyan1'))
-
- ## Warning: Using size for a discrete variable is not advised.
-
- ## Warning: Removed 7 rows containing missing values (geom_point).
वेन्-चित्रस्य उपयोगः भिन्नरूपेण अभिव्यक्तजीनानां विविधसूचीनां मध्ये सम्बन्धानां अन्वेषणार्थं, अथवा कार्यात्मकविश्लेषणेषु बहुमार्गजीनानां प्रतिच्छेदनस्य अन्वेषणार्थं कर्तुं शक्यते वेन् चित्राणि न केवलं अतिव्याप्तजीनानां संख्यां दर्शयन्ति, अपितु जीनस्य अभिव्यक्तिप्रतिमानस्य विषये सूचनां अपि दर्शयन्ति (प्रायः अप-रेगुलेटेड्, प्रायः डाउन-रेगुलेटेड् अथवा प्रति-रेगुलेटेड्) सम्प्रति त्रयः यावत् दत्तांशसमूहाः निवेशरूपेण उपयुज्यन्ते । इनपुट् दत्तांशे न्यूनातिन्यूनं द्वौ स्तम्भौ स्तः: एकः जीननामानां कृते अपरः logFC मूल्यानां कृते ।
- l1 <- subset(circ, term == 'heart development', c(genes,logFC))
- l2 <- subset(circ, term == 'plasma membrane', c(genes,logFC))
- l3 <- subset(circ, term == 'tissue morphogenesis', c(genes,logFC))
- GOVenn(l1,l2,l3, label = c('heart development', 'plasma membrane', 'tissue morphogenesis'))
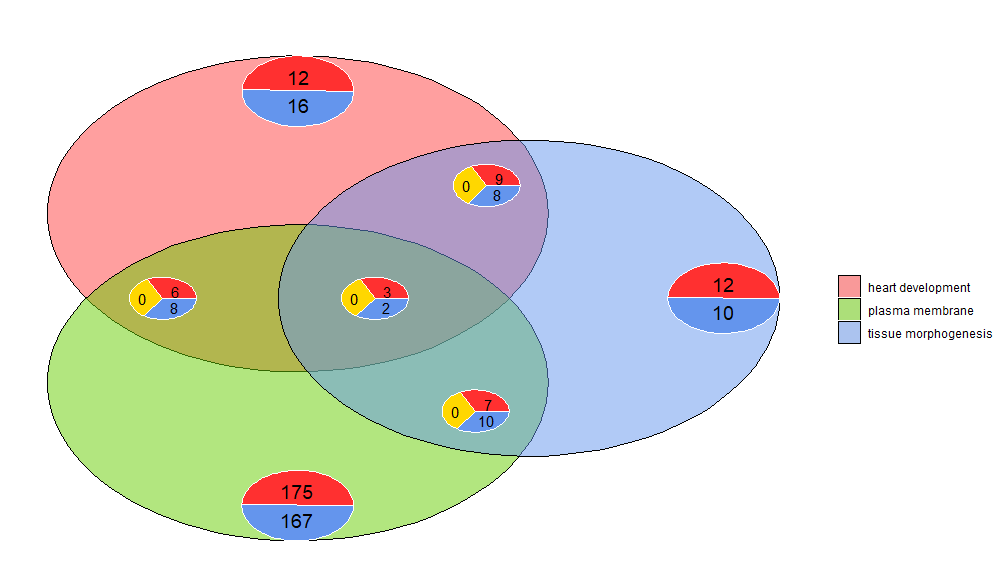
यथा हृदयस्य विकासः ऊतकरूपजननं च २२ जीनाः, १२ अप-रेगुलेटेड्, १० डाउन-रेगुलेटेड् च सन्ति । एकं महत्त्वपूर्णं वस्तु अस्ति यत् पाई चार्ट्स् अनावश्यकसूचनाः न प्रदर्शयन्ति । अतः यदि त्रयः दत्तांशसमूहाः तुलना क्रियते तर्हि सर्वेषां दत्तांशसमूहानां (मध्यपाईचार्ट्) सामान्याः जीनाः अन्येषु पाईचार्टेषु न समाविष्टाः भवन्ति । इदं साधनं shinyapp https://wwalter.shinyapps.io/Venn/ इत्यत्र उपलभ्यते, जालसाधनं अधिकं अन्तरक्रियाशीलं भवति, वृत्तस्य क्षेत्रफलं दत्तांशसमूहे जीनसङ्ख्यायाः आनुपातिकं भवति, स्लाइडरस्य उपयोगेन च... small pie chart, and has GOVenn Features कथानकस्य विन्यासं परिवर्तयितुं तथा च चित्राणि जीनसूचीं च डाउनलोड् कर्तुं सर्वे विकल्पाः सन्ति ।
सॉफ्टवेयर मुखपृष्ठम् : https://wencke.github.io/